Nat Comput Sci | 分而治之!基于子任务分解的单细胞扰动人工智能模型 STAMP
STAMP方法通过将复杂的基因干扰预测问题分解为三个子任务,展示了与人类认知过程相似的“分而治之”策略。这种方法灵活的借助了当前单细胞扰动领域主流的深度学习模型(如图神经网络、Transformer等),同时其在解决高维数据问题上的思路有相通之处。相较于传统的单一任务模型,STAMP的子任务设计使得其更适应于处理数据维度高且具有稀疏性特征的问题。此外,STAMP与现有的基因调控网络建模方法(如Ce
生信碱移
STAMP: 单细胞扰动预测
单细胞遗传扰动的转录结果揭示了细胞功能以及细胞如何响应外部干预。各种高通量测序技术,包括单细胞 RNA 测序和单细胞规律间隔短回文重复(CRISPR)遗传筛选方法(例如,Perturb-seq和CROP-seq)。尽管通过高通量测序可以生成单个基因或细胞中多个基因的遗传干扰结果,但由于基因的组合可以非常多,粗暴的实验探索多基因扰动的结果是不切实际的。此外,单细胞 CRISPR 基因筛选技术仍处于早期阶段且成本效益低,获得的细胞系扰动数据有限。因此,迫切需要开发计算方法,以指导在涉及单基因扰动、多基因扰动和跨细胞系适应的各种场景中预测遗传扰动后的转录变化。

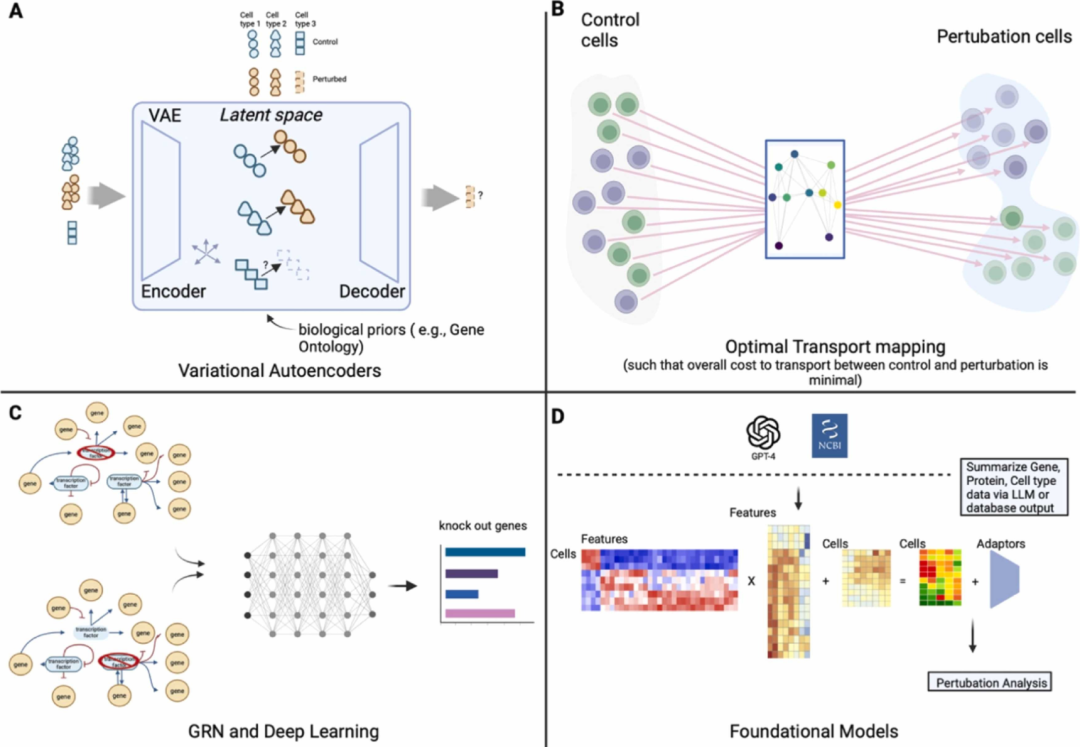
▲ 图 :单细胞生物学中扰动建模的现状。(A) 变分自编码器(VAEs)预测在训练数据中缺失的未见扰动,以进行潜在空间学习,同时可以添加符合生物学过程的可解释性架构,比如ontoVAE;(B) OT 映射预测来自未配对对照测量的扰动细胞的分布;(C) 基因调控网络(GRN)启发的模型为潜在关键转录因子的虚拟敲除提供了机会,利用神经网络的非线性外推来辨别哪些基因受到更大影响;(D) 单细胞扰动建模的最新趋势从大型单细胞基础模型中汲取灵感,结合来自LLM模型的嵌入和生物信息学数据库与单细胞组学,以制定扰动预测。图片来源:10.1016/j.csbj.2024.04.058。
为此,来自同济大学介绍了一种基于子任务分解(Subtask Decomposition Modeling)的人工智能方法STAMP,于2024年9月27日发表于Nature Computational Science [IF:12.0],用于预测基因干扰的结果。这种方法将扰动预测任务的根本性质解释为三个子任务,即找到可以被干扰强烈影响的基因、基因的表达变化方向以及表达变化程度。作者的研究结果展现出STAMP在不同数据集和细胞系中的预测能力的显著提升,尤其是在小样本情况下也能有效识别关键调控基因和通路,并揭示多种基因间复杂的相互作用。

▲ DOI: 10.1038/s43588-024-00698-1
STAMP架构
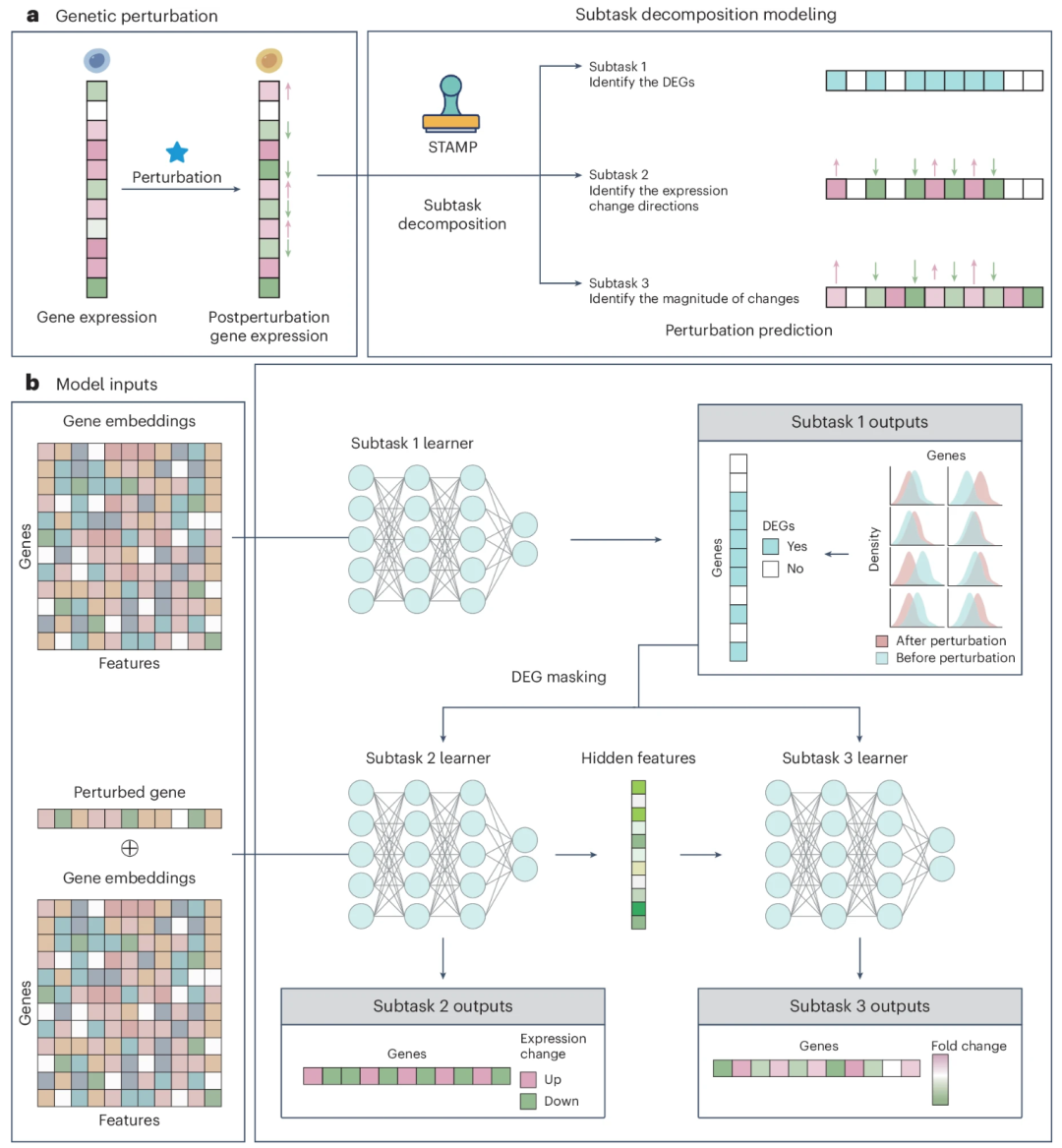
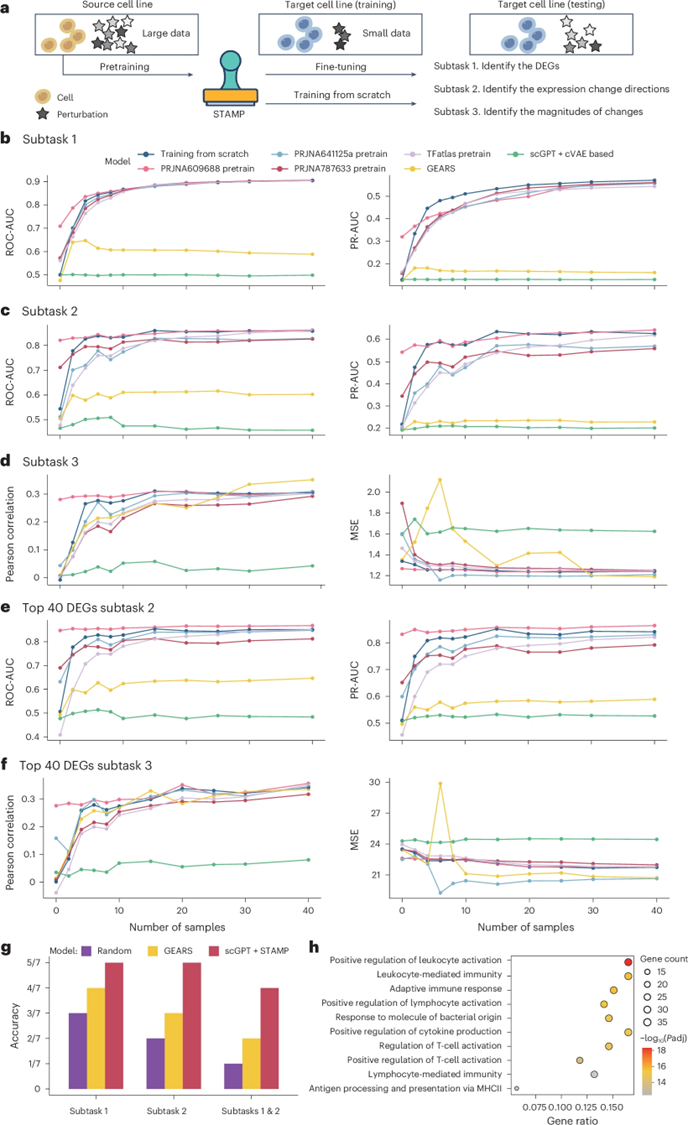
STAMP 接收一个包含多个细胞的基因表达和扰动信息的数据集,利用预训练的基因嵌入模型(如 scBERT、Geneformer),将每个基因转化为低维嵌入向量。对于每个细胞的扰动集合,模型通过对所有基因嵌入取均值生成扰动嵌入,然后将该扰动嵌入与基因嵌入进行拼接,形成组合特征。通过学习基因在不同扰动条件下的表现,STAMP 通过以下顺序的子任务进行扰动预测:
-
子任务1:识别扰动后的差异表达基因(DEGs)。
-
子任务2:确定这些 DEGs 在扰动后的表达变化方向(上调或下调)。
-
子任务3:定量分析这些 DEGs 的表达变化幅度。
STAMP的应用
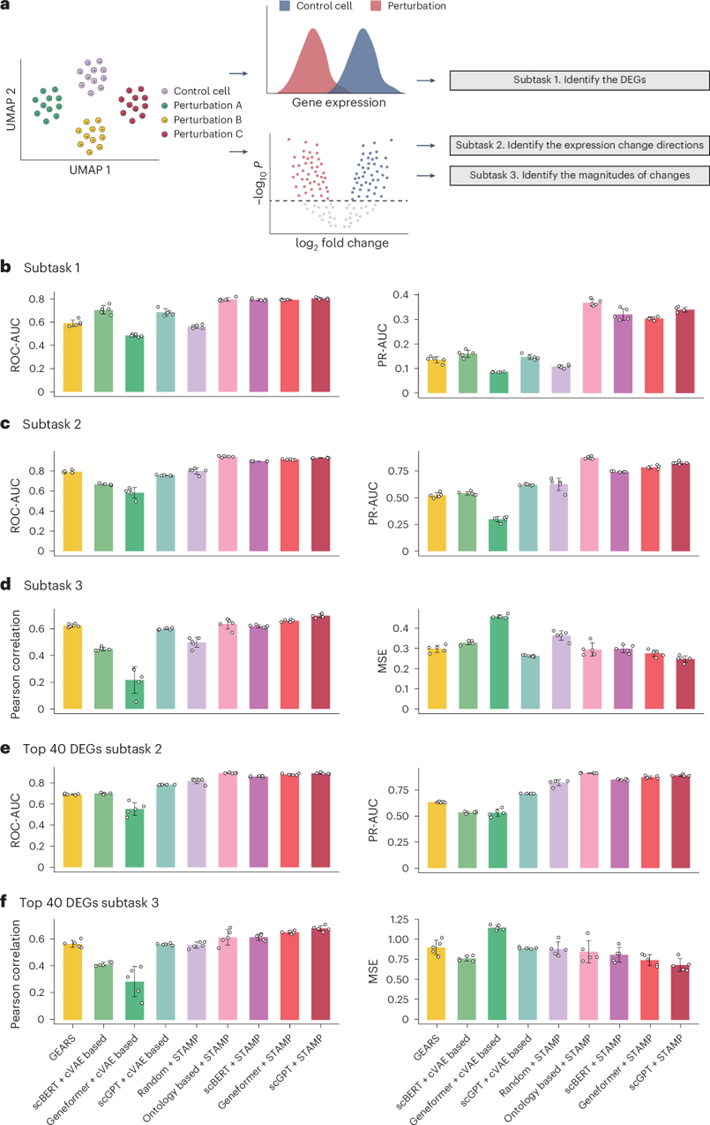
预测单基因扰动结果
预测多基因扰动结果
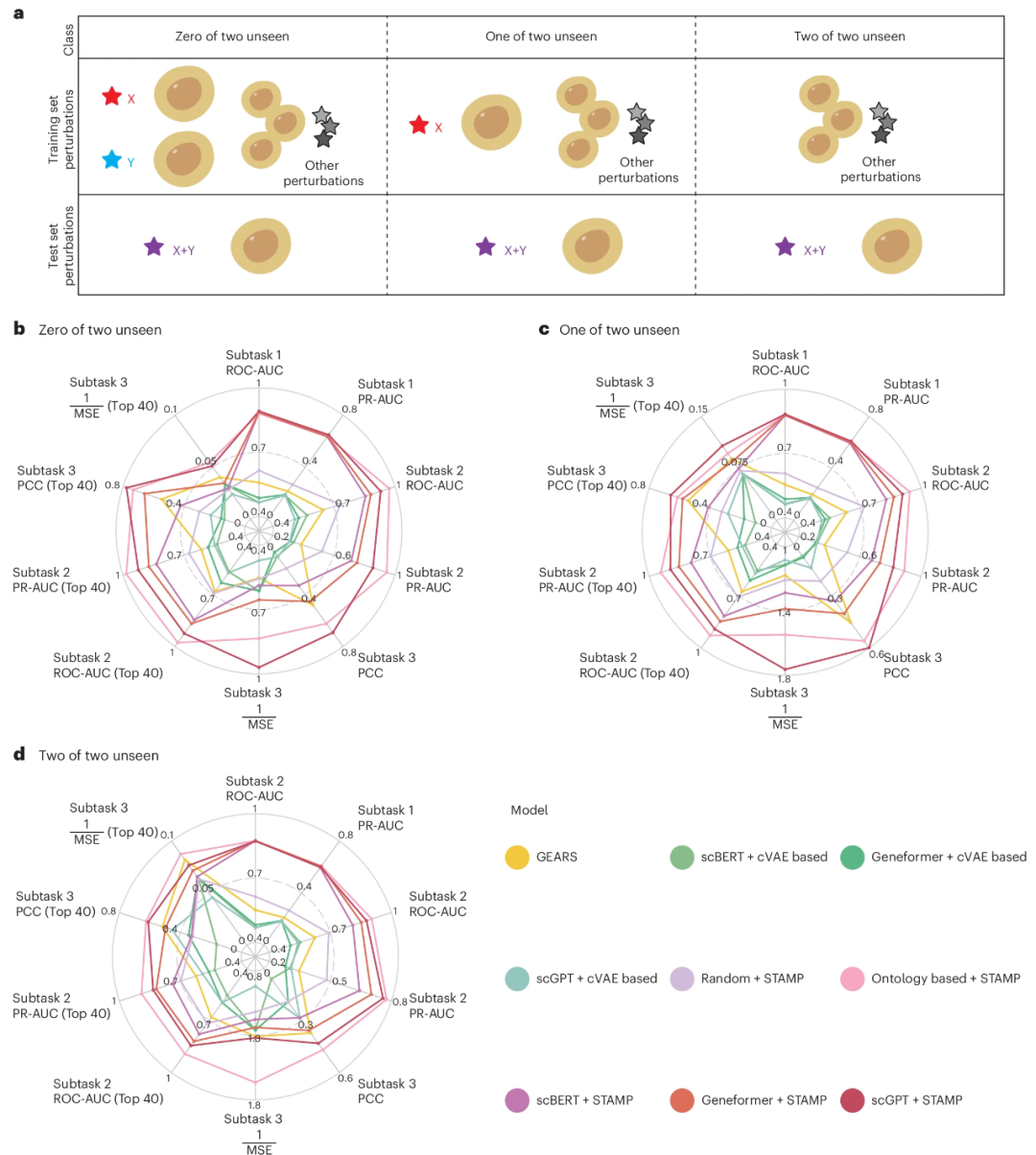
预测不同细胞系的基因扰动结果
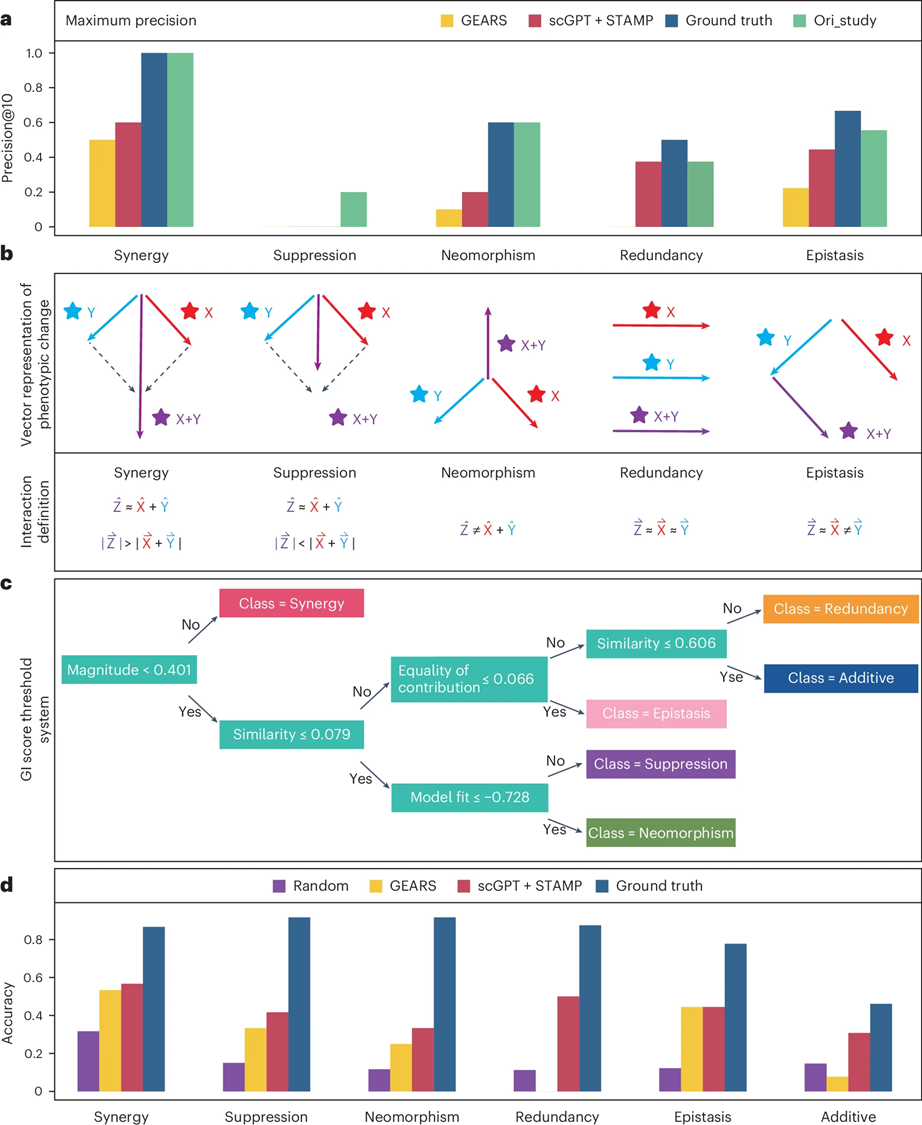
基因交互(GI)类型识别结果
总结
STAMP方法通过将复杂的基因干扰预测问题分解为三个子任务,展示了与人类认知过程相似的“分而治之”策略。这种方法灵活的借助了当前单细胞扰动领域主流的深度学习模型(如图神经网络、Transformer等),同时其在解决高维数据问题上的思路有相通之处。相较于传统的单一任务模型,STAMP的子任务设计使得其更适应于处理数据维度高且具有稀疏性特征的问题。此外,STAMP与现有的基因调控网络建模方法(如CellOracle和SCENIC+)相比,提供了更为灵活的嵌入策略,有望为研究复杂的基因调控机制提供新视角。

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容





所有评论(0)