GA遗传算法-Python实现
使用遗传算法(GA)解决函数求最大值问题
文章目录
前言
善始者繁多,克终者盖寡。
GA(Genetic Algorithm)译为遗传算法,以达尔文进化理论为基础,模拟生物进化过程以获取问题最优解的算法。GA算法可以用来处理最值问题,在神经网络中也有广泛应用。
一、问题描述——求函数最大值
现要求函数在[0,10]内的最大值,函数为:

使用matplotlib绘制其图像

有人会说,对于这样的求最大值问题,直接穷举,挨个把x带入求出最大值不就好了吗?
对于当前问题当然是可行的,因为此时的输入较小,其范围为[0,10],若我们要求解[0,999999]间的最大值呢?此时用穷举就太过费时费力了!!!
二、遗传算法(GA)
2.1 工作原理
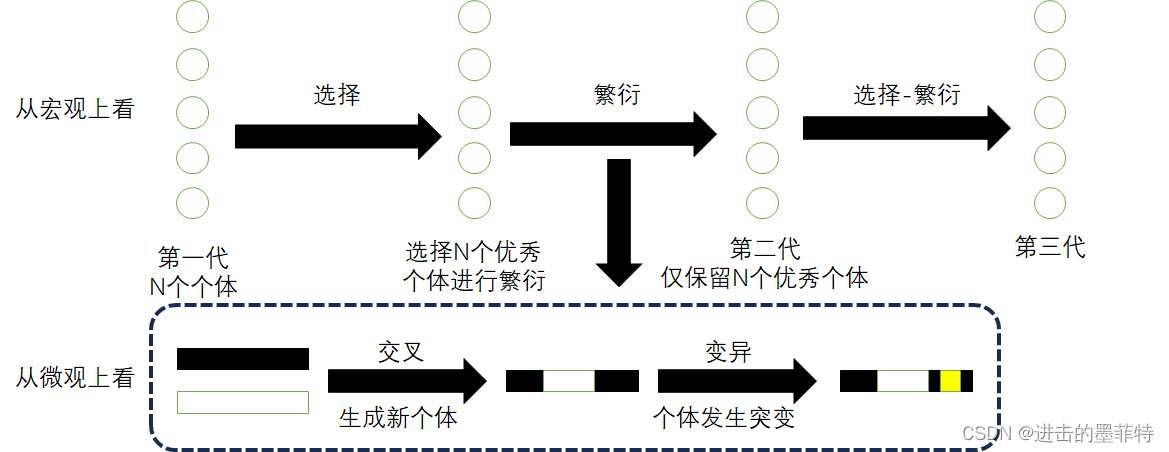
GA模拟了生物的进化过程,更为形象的例子是孟德尔的水稻杂交实验。我们用图示法来理解:

从宏观上看,GA是对种群进行“选择”和“繁衍”操作,在所有个体中挑选指定数量的优秀个体生成新的种群;
从微观上看,“繁衍”操作是生物DNA的改变,包含染色体的“交叉”和“变异”两个操作。
使用流程图可以表述为:

2.2 名词解释
生物繁衍的流程我们已经清楚,但我们还需要解决如下问题:
种群中的个体如何表示?
如果判断个体更优秀?
如何挑选更优秀的个体进行繁衍?
交叉如何实现
个体变异如何实现
2.2.1 编码——个体的表示
我们需要求第一章中函数的最大值,函数的输入是[0,10]间的有理数,可以是1,2,3这种整数,还可以是1.5,5.5这样的小数,为方便理解我们现仅考虑整数的表示(代码中是不存在此问题)。
最直接的方法时使用二进制数表示输入数据的编码。输入空间中最大值为10,所有我们使用4为二进制数就可以表示[0,10]内的所有输入:
2表示为0010;
8表示为1000;
10表示为1010.
使用二进制数表示输入的目的是:方便交叉和变异操作。通常情况下编码的长度为10或20,编码的长度越长越好,但是随着编码长度的增加,计算量也随之增大,本文中编码的长度设置为10。
但是,我们现在最大的输入才是10,10个长度的编码最大可以表示为2^10-1,即1023,例如,此时有:
2的编码表示为0000000010
3的编码表示为0000000011
这么多的空间都浪费掉了,将原问题中[0,10]作为输入不可取。
所有我们直接将10为二进制数作为输入,再经变换将其映射到区间[0,10]内,其公式为:
BtoD(x)表示将输入的二进制串转换为十进制数;
2^len-1表示当前二进制编码能够表示的最大值,本文中len值为10,其表示的最大值为1023;
UP表示当前输入域的最大值,本文中输入范围为[0,10],所有此时UP值为10
例如输入1111111111,表示在函数中输入的x值为10;
例如输入0000000001,表示在函数中输入的x值为0.009775171065493646;
例如输入0000000000,表示在函数中输入的x值为0。
2.2.2 适合度——判断哪个个体更优秀
适合度描述了当前个体在当前问题的适合情况,个体的适合度越高说明该个体越优秀,那么在选择和繁衍时应当有较大概率将其保留下来。
用来计算适合度的函数就是适合度函数。 适合度函数的选择需要根据具体问题制定,本文拟求解函数的最大值,那么使用原函数即可,为保证个体的适合度不为负值,其计算公式为:

fitness表示个体的适合度得分;
data表示该个体在原函数上的计算值。
2.2.3 轮盘赌选择法——选择更优秀个体
“轮盘赌选择法”是一个文绉绉的叫法,简单理解:适合度更高的个体被留下的概率更高,适合度低的个体被剔除的概率更高。

现种群中有4个个体,分别计算出其适合度后计算每个个体适合度的比例,其中D个体的适合度比例最高,它被留下的概率最大,C被留下的概率最小,python中通过random函数实现。
个体适合度比例的计算公式为:

适合度比例p在后续操作中也被作为“概率”使用。
2.2.4 交叉——生成新个体
通过将两个二进制序列进行交叉,可以得到一个新的序列,我们成原来的两个二进制序列为parent,成新生成的二进制序列为child。那么问题又来了:
在哪个位置交叉呢? 交叉位置随机选择。
交叉多少呢? 交叉位数同样随机选择。
看个例子:

2.2.5 变异——增加样本输入空间
变异过程模拟了生物中的遗传与变异中的“变异”,具体操作就是改变一个二进制序列随机位置的值,将0换为1,将1换为0。变异的概率对程序的运行结果有很大的影响,通常情况下变异概率的取值范围为1%~10%。本文中变异概率取1%。
2.3 工作流程
网络中关于GA的工作流程大多描述为:
 在此基础上我们进行改进,得到如下流程图:
在此基础上我们进行改进,得到如下流程图:

整个流程描述为:输入种群数据(二进制序列)后,将其映射到输入空间,计算个体适合度,将适合度比例转换为“概率”挑选出优秀的个体,再次计算种群中个体的适合度,因为我们有理由让优秀的个体繁衍的概率更大,最后进行交叉、变异操作生成新的种群,重复上述操作直至繁衍结束。
注意:不能够按照适合度大小来挑选适合度高的个体,假设A个体适合度10,B个体适合度10.1,我们不能够人为挑选B,因为说不定经过变异,B的适合度就高于A了,此处我们都以概率的方式决定个体的去留。
三、python代码
3.1 目标函数
def fx(x):
return numpy.sin(10*x)*x+numpy.cos(2*x)*x
# return x*x*x*3+x*x*2
# return 10*numpy.sin(5*x)+7*numpy.cos(4*x)
3.2 进制转换
def otob(data):
temp = []
while data!=0:
temp.append(data%2)
data = int(data/2)
temp.reverse()
return temp
#DNA翻译,讲二进制转换为十进制并将其缩放到(0,10)内
def btoo(data):
sum = 0
for i in range(len(data)):
sum = sum + data[i]*2**(len(data)-i-1)
return sum/(2**DNA_SIZE-1)*10
3.3 适合度函数
为了保证程序的正常运行,在计算结果是加上一个极小值。
#定义适合度函数,此时使用原数值即可
def fitness(data):
return data-numpy.min(data)+1e-5
3.4 选择
temp 操作表示将适合度得分转换为“概率”。此时temp为二维数组,需将其转换为一维数组。
#适者生存,从种群中挑选出指定数量的优质个体(依据适合度)
'''
replace=True表示可以取出相同的值,默认为False
'''
def select(pop,fitness_score):
temp = fitness_score/numpy.sum(fitness_score)
p = []
for data in temp:
p.append(data[0])
#轮渡算法,按照概率挑选个体
index = numpy.random.choice(numpy.arange(POP_SIZE),size=POP_SIZE,replace=True,p=p)
return pop[index]
3.5 交叉
交叉操作同样依据适合度来选择parent,因为我们有理由让适合度高的个体有更大的概率参与繁衍。
#繁衍,父母DNA配对生成新的个体(新的个体会替代原来的个体),通过交叉功能实现,交叉位置随机
def cross_fitness(pop,fitness_score):
temp = fitness_score / numpy.sum(fitness_score)
p = []
for data in temp:
p.append(data[0])
#优秀的个体其繁衍概率应该更大
parent1_index = numpy.random.choice(numpy.arange(POP_SIZE),p=p)
parent2_index = numpy.random.choice(numpy.arange(POP_SIZE),p=p)
parent1 = pop[parent1_index]
parent2 = pop[parent2_index]
if numpy.random.rand() <= CROSS_RATION:
#两个体在随机位置交叉,生成新的个体,此处使用True和False数组表示
cross_index = numpy.random.randint(0, 2, size=DNA_SIZE).astype(numpy.bool_)
parent1[cross_index] = parent2[cross_index]
return parent1
3.6 变异
变异操作相对简单,只需将对应位置的0和1即可。
#变异,在繁衍过程中有小概率出现变异的情况
def mutation(child):
for i in range(DNA_SIZE):
if numpy.random.rand() <= MUTATION_RATION:
if child[i] == 0:
child[i] = 1
else:
child[i] = 0
return child
四、程序测试
4.1 测试程序
依据2.3中工作流程初始化种群并进行相应操作,最后使用matplotlib绘制图像。
if __name__ == '__main__':
#生成初始种群
pop = numpy.random.randint(0,2,size=(POP_SIZE,DNA_SIZE))
# print(pop)
#繁衍GENERATION次
for i in range(GENERATION):
#讲二进制转换为十进制
pop_value = []
for j in range(POP_SIZE):
temp = []
temp.append(btoo(pop[j]))
pop_value.append(temp)
# print(pop_value)
#计算个体适合度得分,此问题中希望fx的取值尽量大,此时就通过fx的值计算适合度
pop_value_fitness = []
for temp_data in pop_value:
temp = []
temp.append(fx(temp_data[0]))
pop_value_fitness.append(temp)
pop_value_fitness = fitness(pop_value_fitness)
# print(pop_value_fitness)
#适者生存,从中群众挑选出优质的个体
pop = select(pop,pop_value_fitness)
# 讲二进制转换为十进制
pop_value = []
for j in range(POP_SIZE):
temp = []
temp.append(btoo(pop[j]))
pop_value.append(temp)
# print(pop_value)
# 计算个体适合度得分,此问题中希望fx的取值尽量大,此时就通过fx的值计算适合度
pop_value_fitness = []
for temp_data in pop_value:
temp = []
temp.append(fx(temp_data[0]))
pop_value_fitness.append(temp)
pop_value_fitness = fitness(pop_value_fitness)
#繁衍,种群中的个体进行配对
new_pop = []
# for parent in pop:
# child = cross(parent,pop)
# #在繁衍的过程中子代可能出现变异
# child = mutation(child)
# #将新产生的个体重新放回种群
# new_pop.append(child)
for i in range(POP_SIZE):
child = cross_fitness(pop,pop_value_fitness)
child = mutation(child)
new_pop.append(child)
pop = numpy.array(new_pop)
# 绘制原始曲线
data = []
xindex = []
i = 1
while i <= 10:
data.append(fx(i))
xindex.append(i)
i += 0.1
pyplot.plot(xindex, data)
# 绘制经过N次繁衍后剩下种群散点分布
# print(pop)
data2 = []
xindex2 = []
for i in range(len(pop)):
xindex2.append(btoo(pop[i]))
data2.append(fx(btoo(pop[i])))
print(xindex2)
pyplot.scatter(xindex2, data2, color='red')
pyplot.show()
print(f"最大值为{numpy.max(data2)}")
4.2 运行结果
我们发现绝大多数点的x坐标集中在9.5附近,已经覆盖了最大值附近范围,同时图中还有一些不应当出现的点,程序还可以继续优化。


五、完整代码
import numpy
from matplotlib import pyplot
# 基因序列长度
DNA_SIZE = 10
# 初始种群数量
POP_SIZE = 100
# 交叉配对的概率
CROSS_RATION = 0.5
# 变异概率
MUTATION_RATION = 0.01
#繁衍代数
GENERATION = 500
def fx(x):
return numpy.sin(10*x)*x+numpy.cos(2*x)*x
# return x*x*x*3+x*x*2
# return 10*numpy.sin(5*x)+7*numpy.cos(4*x)
def draw_primal():
data = []
xindex = []
i = 1
while i <= 10:
data.append(fx(i))
xindex.append(i)
i+=0.1
pyplot.plot(xindex,data)
pyplot.show()
def otob(data):
temp = []
while data!=0:
temp.append(data%2)
data = int(data/2)
temp.reverse()
return temp
#DNA翻译,讲二进制转换为十进制并将其缩放到(0,10)内
def btoo(data):
sum = 0
for i in range(len(data)):
sum = sum + data[i]*2**(len(data)-i-1)
return sum/(2**DNA_SIZE-1)*10
#归一化处理
def guiyi(data):
data = (data-numpy.min(data))/(numpy.max(data)-numpy.min(data))
return data
#定义适合度函数,此时使用原数值即可
def fitness(data):
return data-numpy.min(data)+1e-5
#适者生存,从种群中挑选出指定数量的优质个体(依据适合度)
'''
replace=True表示可以取出相同的值,默认为False
'''
def select(pop,fitness_score):
temp = fitness_score/numpy.sum(fitness_score)
p = []
for data in temp:
p.append(data[0])
#轮渡算法,按照概率挑选个体
index = numpy.random.choice(numpy.arange(POP_SIZE),size=POP_SIZE,replace=True,p=p)
return pop[index]
#繁衍,父母DNA配对生成新的个体(新的个体会替代原来的个体),通过交叉功能实现,交叉位置随机
def cross(parent,pop):
if numpy.random.rand() <= CROSS_RATION:
# 为parent随机寻找一个样本进行配对
index = numpy.random.randint(0, POP_SIZE)
# 在随机位置进行交叉
cross_index = numpy.random.randint(0, 2, size=DNA_SIZE).astype(numpy.bool_)
parent[cross_index] = pop[index][cross_index]
return parent
#繁衍,父母DNA配对生成新的个体(新的个体会替代原来的个体),通过交叉功能实现,交叉位置随机
def cross_fitness(pop,fitness_score):
temp = fitness_score / numpy.sum(fitness_score)
p = []
for data in temp:
p.append(data[0])
#优秀的个体其繁衍概率应该更大
parent1_index = numpy.random.choice(numpy.arange(POP_SIZE),p=p)
parent2_index = numpy.random.choice(numpy.arange(POP_SIZE),p=p)
parent1 = pop[parent1_index]
parent2 = pop[parent2_index]
if numpy.random.rand() <= CROSS_RATION:
#两个体在随机位置交叉,生成新的个体,此处使用True和False数组表示
cross_index = numpy.random.randint(0, 2, size=DNA_SIZE).astype(numpy.bool_)
parent1[cross_index] = parent2[cross_index]
return parent1
#变异,在繁衍过程中有小概率出现变异的情况
def mutation(child):
for i in range(DNA_SIZE):
if numpy.random.rand() <= MUTATION_RATION:
if child[i] == 0:
child[i] = 1
else:
child[i] = 0
return child
if __name__ == '__main__':
#生成初始种群
pop = numpy.random.randint(0,2,size=(POP_SIZE,DNA_SIZE))
# print(pop)
#繁衍GENERATION次
for i in range(GENERATION):
#讲二进制转换为十进制
pop_value = []
for j in range(POP_SIZE):
temp = []
temp.append(btoo(pop[j]))
pop_value.append(temp)
# print(pop_value)
#计算个体适合度得分,此问题中希望fx的取值尽量大,此时就通过fx的值计算适合度
pop_value_fitness = []
for temp_data in pop_value:
temp = []
temp.append(fx(temp_data[0]))
pop_value_fitness.append(temp)
pop_value_fitness = fitness(pop_value_fitness)
# print(pop_value_fitness)
#适者生存,从中群众挑选出优质的个体
pop = select(pop,pop_value_fitness)
# 讲二进制转换为十进制
pop_value = []
for j in range(POP_SIZE):
temp = []
temp.append(btoo(pop[j]))
pop_value.append(temp)
# print(pop_value)
# 计算个体适合度得分,此问题中希望fx的取值尽量大,此时就通过fx的值计算适合度
pop_value_fitness = []
for temp_data in pop_value:
temp = []
temp.append(fx(temp_data[0]))
pop_value_fitness.append(temp)
pop_value_fitness = fitness(pop_value_fitness)
#繁衍,种群中的个体进行配对
new_pop = []
# for parent in pop:
# child = cross(parent,pop)
# #在繁衍的过程中子代可能出现变异
# child = mutation(child)
# #将新产生的个体重新放回种群
# new_pop.append(child)
for i in range(POP_SIZE):
child = cross_fitness(pop,pop_value_fitness)
child = mutation(child)
new_pop.append(child)
pop = numpy.array(new_pop)
# 绘制原始曲线
data = []
xindex = []
i = 1
while i <= 10:
data.append(fx(i))
xindex.append(i)
i += 0.1
pyplot.plot(xindex, data)
# 绘制经过N次繁衍后剩下种群散点分布
# print(pop)
data2 = []
xindex2 = []
for i in range(len(pop)):
xindex2.append(btoo(pop[i]))
data2.append(fx(btoo(pop[i])))
print(xindex2)
pyplot.scatter(xindex2, data2, color='red')
pyplot.show()
print(f"最大值为{numpy.max(data2)}")

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献5条内容
已为社区贡献5条内容






所有评论(0)