GSEAvis富集分析可视化
GseaVis,专门用于GSEA富集分析可视化,相比于enrichplot,增加了很多好用的功能,很多功能让我直呼泰裤辣!!
今天继续介绍富集分析可视化哦~往期推文链接:
今天要介绍的老俊俊的R包:GseaVis,专门用于GSEA富集分析可视化,相比于enrichplot,增加了很多好用的功能,很多功能让我直呼 泰裤辣!!
本期目录:
准备数据
用gse87466这个GEO的数据做演示,下载整理的过程这次就不演示了。数据可在粉丝QQ群免费下载。
library(easyTCGA)
load(file = "G:/easyTCGA_test/gse87466.Rdata")
这是一个炎症性肠病的数据集,一共108个样本,21个normal,87个uc(ulcerative colitis)。
探针注释我已经提前做好了,但是有一些探针对应多个symbol,为了方便我这里直接删掉了:
exprSet[1:4,1:4]
## GSM2332098 GSM2332099 GSM2332100
## IGK@ /// IGKC 13.86197 13.76880 13.95740
## 13.95740 13.92619 13.79664
## IGL@ 13.73797 13.61266 13.86197
## IGH@ /// IGHA1 /// IGHA2 /// LOC100126583 13.79664 13.16844 13.76880
## GSM2332101
## IGK@ /// IGKC 13.95740
## 13.86197
## IGL@ 13.76880
## IGH@ /// IGHA1 /// IGHA2 /// LOC100126583 13.73797
group <- factor(group_list,levels = c("normal","UC"))
table(group)
## group
## normal UC
## 21 87
首先对这个数据做下差异分析,也是用easyTCGA包,1行代码即可,基因芯片数据也是支持的,并且它会自动检测需不需要进行log2转换,如果是count矩阵,会自动使用DESeq2、limma、edgeR进行差异分析,如果不是,会自动进行wilcoxon和limma的差异分析:
library(easyTCGA)
diff_res <- diff_analysis(exprset = exprSet
, group = group
, is_count = F
)
## log2 transform not needed
## => Running limma
## => Running wilcoxon test
## => Analysis done.
# limma的结果
diff_limma <- diff_res$deg_limma
# 多个gene symbol的直接删除,方便演示
diff_limma <- diff_limma[!grepl("/",diff_limma$genesymbol),]
head(diff_limma)
## logFC AveExpr t P.Value adj.P.Val B
## SLC6A14 5.024103 9.413107 21.56440 4.104849e-41 8.514279e-37 82.58182
## LOC389023 -3.550396 5.541681 -21.01057 4.054400e-40 4.204818e-36 80.36199
## SLC23A1 -2.473180 5.649224 -17.88487 3.378001e-34 2.335550e-30 67.08748
## DUOX2 4.911030 9.916299 17.37129 3.569259e-33 1.850839e-29 64.78265
## DPP10 -1.910958 3.991413 -16.98863 2.113068e-32 7.304876e-29 63.04259
## TIMP1 2.125930 11.402645 16.88534 3.425860e-32 1.015131e-28 62.56956
## genesymbol
## SLC6A14 SLC6A14
## LOC389023 LOC389023
## SLC23A1 SLC23A1
## DUOX2 DUOX2
## DPP10 DPP10
## TIMP1 TIMP1
GSEA富集分析
富集分析首选clusterProfiler,没有之一!简单,好用!
clusterProfiler为我们提供了非常好用的ID转换函数,这里的ID转换和上面说的探针注释并不是一回事:
suppressMessages(library(clusterProfiler))
gene_entrezid <- bitr(geneID = diff_limma$genesymbol
, fromType = "SYMBOL" # 从symbol
, toType = "ENTREZID" # 转成ENTREZID
, OrgDb = "org.Hs.eg.db"
)
##
## 'select()' returned 1:many mapping between keys and columns
head(gene_entrezid)
## SYMBOL ENTREZID
## 1 SLC6A14 11254
## 3 SLC23A1 9963
## 4 DUOX2 50506
## 5 DPP10 57628
## 6 TIMP1 7076
## 7 LCN2 3934
富集分析最好用ENTREZID进行,关于多种不同的ID,在曾老师的书中都有详细介绍,强烈推荐初学者一定要看:生信初学者基础知识资源推荐
做GSEA分析对数据格式有要求,之前也说过,需要是一个有序的数值型向量,其名字是基因的ID
gene_entrezid <- merge(gene_entrezid,diff_limma,by.x = "SYMBOL", by.y = "genesymbol")
genelist <- gene_entrezid$logFC
names(genelist) <- gene_entrezid$ENTREZID
genelist <- sort(genelist,decreasing = T)
head(genelist)
## 4314 11254 50506 1673 1116 6279
## 5.123666 5.024103 4.911030 4.608619 4.552790 4.256463
我们使用msigdbr包从msigdb数据库下载人类的C5注释集,大家常用的GO、KEGG的数据其实都是包括在msigdb数据库中的。
library(msigdbr)
m_t2g <- msigdbr(species = "Homo sapiens", category = "C5") %>%
dplyr::select(gs_name, entrez_gene)
head(m_t2g)
## # A tibble: 6 × 2
## gs_name entrez_gene
## <chr> <int>
## 1 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS 60496
## 2 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS 10840
## 3 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS 160428
## 4 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS 4522
## 5 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS 25902
## 6 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS 441024
然后是进行GSEA分析:
gsea_res <- GSEA(genelist,
TERM2GENE = m_t2g,
minGSSize = 10,
maxGSSize = 500,
pvalueCutoff = 0.05,
pAdjustMethod = "BH",
seed = 456
)
## preparing geneSet collections...
## GSEA analysis...
## leading edge analysis...
## done...
gsea_res_symbol <- setReadable(gsea_res,"org.Hs.eg.db",keyType = "ENTREZID")
富集分析得到的结果是一个对象,关于这个对象包括哪些东西,如何对它进行各种操作,我们在之前的推文都介绍过了,这里就不多说了~
GseaVis包的功能很强大。
library(GseaVis)
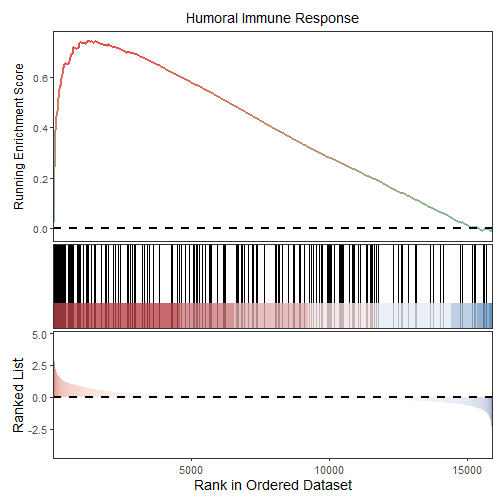
彩色RES图
Running Enrichment Score的线条变成了彩色,注意geneSetID只能接受字符串,不可以直接使用数字。
gseaNb(gsea_res, geneSetID = "GOBP_HUMORAL_IMMUNE_RESPONSE")

通路名截断
还会很贴心的帮你把下划线去掉:
gseaNb(gsea_res,
geneSetID = "GOBP_ANTIMICROBIAL_HUMORAL_IMMUNE_RESPONSE_MEDIATED_BY_ANTIMICROBIAL_PEPTIDE",
termWidth = 30
)

新的gene rank图
彩色的RES以及gene rank图形:
gseaNb(gsea_res,
geneSetID = 'GOBP_HUMORAL_IMMUNE_RESPONSE',
newGsea = T,
addPoint = T,
newHtCol = c("#336699", "white", "#993399")
)

添加NES信息
enrichplot需要自己DIY才能添加NES信息,这里提供了另一种思路:
gseaNb(gsea_res,
geneSetID = 'GOBP_HUMORAL_IMMUNE_RESPONSE',
addPval = T,
pCol = "steelblue",
pvalX = 0.65, # 位置
pvalY = 0.7,
pHjust = 0, # 对齐方式
nesDigit = 4, # 小数点位数
pDigit = 4
)

多个gseaplot拼图
enrichplot的gseaplot2函数的结果是gglist,所以不能直接拼图,但是GseaVis可以:
terms <- gsea_res@result$ID[1:4]
gseaplot_list <- lapply(terms, function(x){
gseaNb(object = gsea_res,
geneSetID = x,
termWidth = 30,
addPval = T,
pvalX = 0.75,
pvalY = 0.6
)
})
# 可以直接拼
cowplot::plot_grid(plotlist=gseaplot_list, ncol = 2)

多条通路另类展示
不得不说颜值上升了好几个高度!
terms <- gsea_res@result$ID[1:3]
gseaNb(object = gsea_res,
geneSetID = terms,
subPlot = 2,
termWidth = 35,
legend.position = c(0.8,0.8),
addPval = T,
pvalX = 0.05,pvalY = 0.05)

dotplot
可以分开展示上调和下调的通路。
dotplotGsea(data = gsea_res,
topn= 10,
str.width = 20 # 折叠通路名,我这里不能用...
)

还可以画成棒棒糖图:
dotplotGsea(data = gsea_res,
topn= 10,
order.by = "NES",
add.seg = T,
line.col = 'orange',
line.type = 1
)

火山图
火山图展示GSEA的结果,泰裤辣!再也不用自己提取结果了。
volcanoGsea(data = gsea_res,
nudge.y = c(-0.8,0.8)
)

添加热图
提供表达矩阵即可,但是第一列需要是基因名字!
library(tibble)
exprset <- rownames_to_column(exprSet,var = "gene")
exprset[1:4,1:4]
## gene GSM2332098 GSM2332099 GSM2332100
## 1 IGK@ /// IGKC 13.86197 13.76880 13.95740
## 2 13.95740 13.92619 13.79664
## 3 IGL@ 13.73797 13.61266 13.86197
## 4 IGH@ /// IGHA1 /// IGHA2 /// LOC100126583 13.79664 13.16844 13.76880
无需手动匹配基因,会自动帮你进行整理:
gseaNb(object = gsea_res_symbol,
geneSetID = 'GOBP_HUMORAL_IMMUNE_RESPONSE',
add.geneExpHt = T,
exp = exprset)

对接GSEA桌面软件的结果
gseaNb 可以直接传入输出文件路径 (filePath) 自动进行分析, 你也可以先使用 readGseaFile 函数读取,再传给 filePath 参数,这样画图就不用每次重新分析整合数据了。
我并不用那个GSEA的软件,所以这里就不展示了~大家可以参考俊俊的推文:GseaVis 一键对接 GSEA 软件结果并可视化
参考资料
大家可以去老俊俊的github查看更加详细的内容:https://github.com/junjunlab/GseaVis
给他点个小星星~

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献38条内容
已为社区贡献38条内容





所有评论(0)