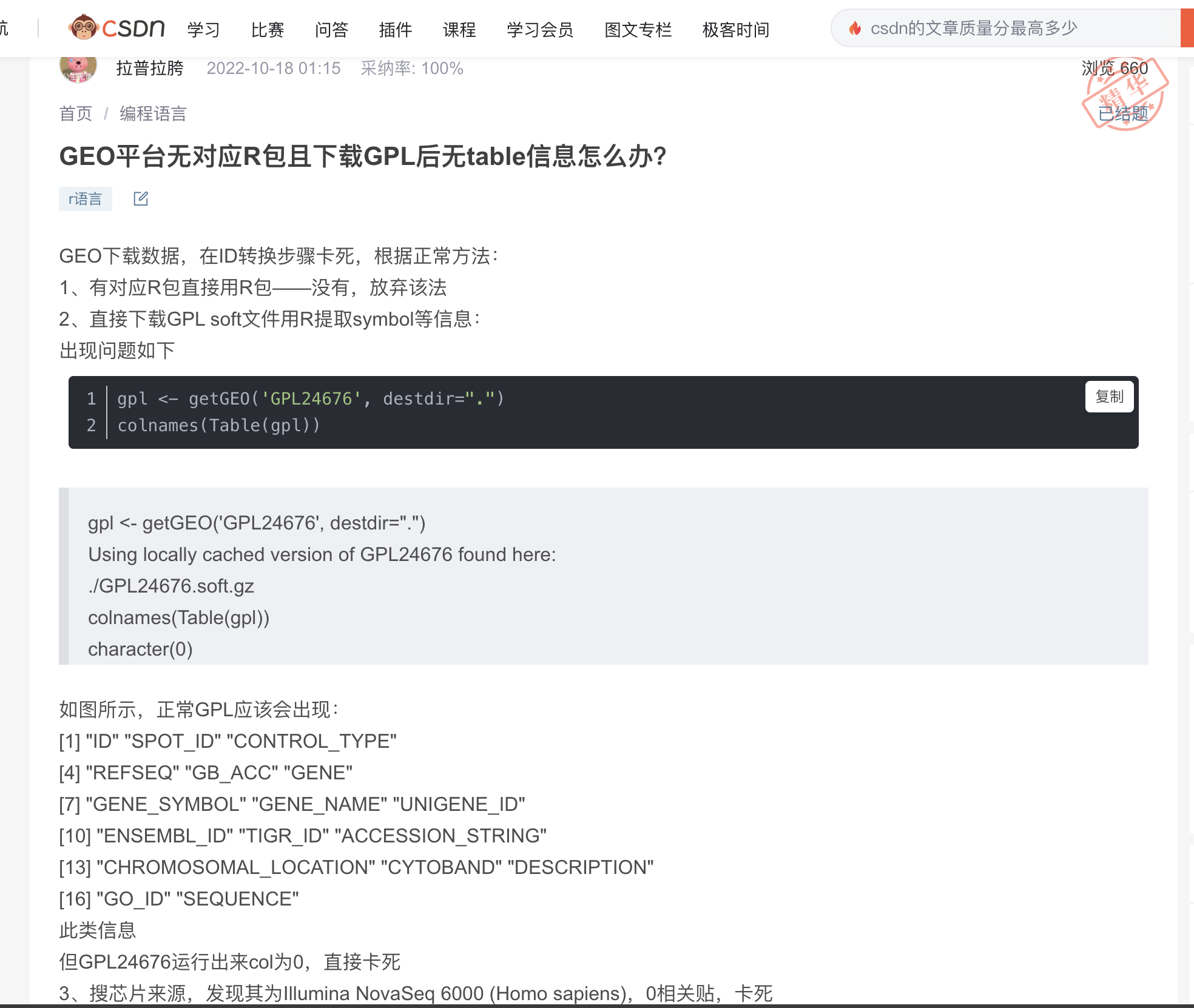
代码分享|GPL平台没有基因注释什么办?别慌,基因ID注释万能公式!
前因是小编在接近两年前回复了C站小伙伴一条帖子,这一年多来陆续有20几个问题,同样是问GPL没有基因注释文件怎么转换Symbol ID说实话我也不知道,如果是做大队列的话一般为了省事我直接换一个GSE,但如果这个数据集真的很好,含泪也要想办法去搞定。而且第一时间看到soft里无symbol或者GPL一般情况下,作者都会在补充文件上传已经注释好ID的表达矩阵,或者把注释文件,直接下载使用即可。第二般
文章目录
1.前言

前因是小编在接近两年前回复了C站小伙伴一条帖子,这一年多来陆续有20几个问题,同样是问GPL没有基因注释文件怎么转换Symbol ID
说实话我也不知道,如果是做大队列的话一般为了省事我直接换一个GSE,但如果这个数据集真的很好,含泪也要想办法去搞定。而且第一时间看到soft里无symbol或者GPL是空的不要慌:
- 一般情况下,作者都会在补充文件上传已经注释好ID的表达矩阵,或者把注释文件,直接下载使用即可。
- 第二般情况下,在谷歌检索这个GSE+symbol,或者检索GPL+gene,或者其中一个id+ann/symbol/gene,比较热门的平台网上都会有大神提供的文件或者结果。常见的都是在github上的
- 第三般情况,真的很新很新这个平台,上官网,Agilent的去Agilent,Illumina的去Illumina,发邮件联系,祝你好运。
- 最后情况,见招拆招,歪门邪道,完全看经验,归根结底还是需要知道这是什么ID,才能去找到对应的注释。小编在后面分享一下解决方案吧。
代码已打包,公众号多线程核糖体后台回复geo转换id即可领取
2.GPL空了怎么办
2.1 google/官网
这两种情况比较繁琐且特殊,没有找到太好的例子去示范,就按上面说的方法去查即可。
2.2 GSE164011
这是第一种情况,GPL21697、GPL24676、GPL29487空空如也

但是看一下补充文件,其实作者已经做了一个转换完ID的矩阵了,留意一下每个sheet,有原始reads的有靶点的还有蛋白的,根据自己需求,一般拿raw_count最就行了

2.3 GSE213001
这个GSE的GPL也是空无一物,不过作者也上传了表达矩阵上来,但是

但是是EntrezID,是的,也不算Symbol ID

不过做过TCGA的小伙伴应该能GET到,这个拿TCGA官网的ann文件转换即可,所以还是熟能生巧,经验法。

2.4 GSE212067(看漏眼情况)
这种不过多阐述了,提问的小伙伴应该看漏了,
gene_assignment这列里有Symbol ID。简单点用excel分列提取可以,这个在R用正则表达式提取第二个//和第三个//之间的内容即可,可以用sub或者gsub,用stringr也行:
library(stringr)
df$ann <- str_extract(df$gene_assignment, "(?<=// ).+?(?= //)")

2.5 GSE242881(还是看漏眼)
这种也是空GPL的,不过补充文件有表达矩阵,先下载

不要看到第一列Ensemble ID就开始找文件,其实Symbol ID也在里面,稍微往后喵点,gene_name这列,这是老鼠的基因所以小写

2.6 GSE146621
这个GEO数据集依旧是无平台注释文件,google也搜不到,官网也不好找,这种怎么办呢。其实做多了GEO的可以一眼看出这个NM_,这个其实是RefSeq,这是NCBI给基因/蛋白做的标识探针靶向的基因序列

关于RefSeq转Symbol网上好像有标准文件的,这里偷了个懒,直接拿个之前带有NM的注释文件,提取了RefSeq和Symbol拿来merge

看了下有点牵强,能转换大概一万两千个左右,能用但不算太好,因为去完重后估计还得少一大段,所以很不推荐

上面是错误示范,标准答案:
还是用org.Hs.eg.db包来注释吧,试了一下,全部都能转换成功,一共35064个,说明还是术业有专攻,这个懒偷不得。。。。
> library('org.Hs.eg.db')
> columns(org.Hs.eg.db)
[1] "ACCNUM" "ALIAS" "ENSEMBL" "ENSEMBLPROT" "ENSEMBLTRANS" "ENTREZID"
[7] "ENZYME" "EVIDENCE" "EVIDENCEALL" "GENENAME" "GENETYPE" "GO"
[13] "GOALL" "IPI" "MAP" "OMIM" "ONTOLOGY" "ONTOLOGYALL"
[19] "PATH" "PFAM" "PMID" "PROSITE" "REFSEQ" "SYMBOL"
[25] "UCSCKG" "UNIPROT"
> length(keys(org.Hs.eg.db, keytype = 'REFSEQ'))
[1] 481819
> ids <- select(org.Hs.eg.db, keys=data$ID, columns = 'SYMBOL', keytype = 'REFSEQ')
> colnames(ids) <- c("ID","Symbol")
> write.table(ids,"ids_all.txt",sep = "\t",row.names = F,quote = F)
> genes <- intersect(data$ID,ids$ID)
> length(genes)
[1] 35064
3.转换ID代码分享链接
此外还有之前分享过的用注释文件转换基因ID的标准流程:
公众号多线程核糖体后台回复geo转换id即可领取项目代码及文件

里面还包含了人源lncRNA、mRNA和miRNA的基因信息汇总文件,方便大家根据研究需求做筛选和过滤

- 如果还有其他难处理的或找不到注释文件的数据集留言讨论

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献10条内容
已为社区贡献10条内容





所有评论(0)