蛋白结构预测RoseTTAFold2的安装及使用
本文介绍了蛋白结构预测开源程序RoseTTAFold2的安装及使用体验。本文介绍了蛋白结构预测开源程序RoseTTAFold2的安装及使用体验,并在实例上与AlphaFold2和ESMFold的表现与耗时做了比较。
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
文章目录
前言
本文介绍了蛋白结构预测开源程序RoseTTAFold2的安装及使用体验。
一、RoseTTAFold2是什么?
github代码:https://github.com/uw-ipd/RoseTTAFold2
介绍文章:https://www.biorxiv.org/content/10.1101/2023.05.24.542179v1.full
RoseTTAFold(RF)1是在AlphaFold2(AF2)2的架构公开之前开发的。虽然这两个模型在发布时都比最先进的蛋白质结构预测有了显著的提高,但AF2总体上具有高得多的准确性。RF和AF2在结构和训练方案上都不同。
作者试图开发一种结合两者特征的改进方法,命名为RoseTTAFold 2。
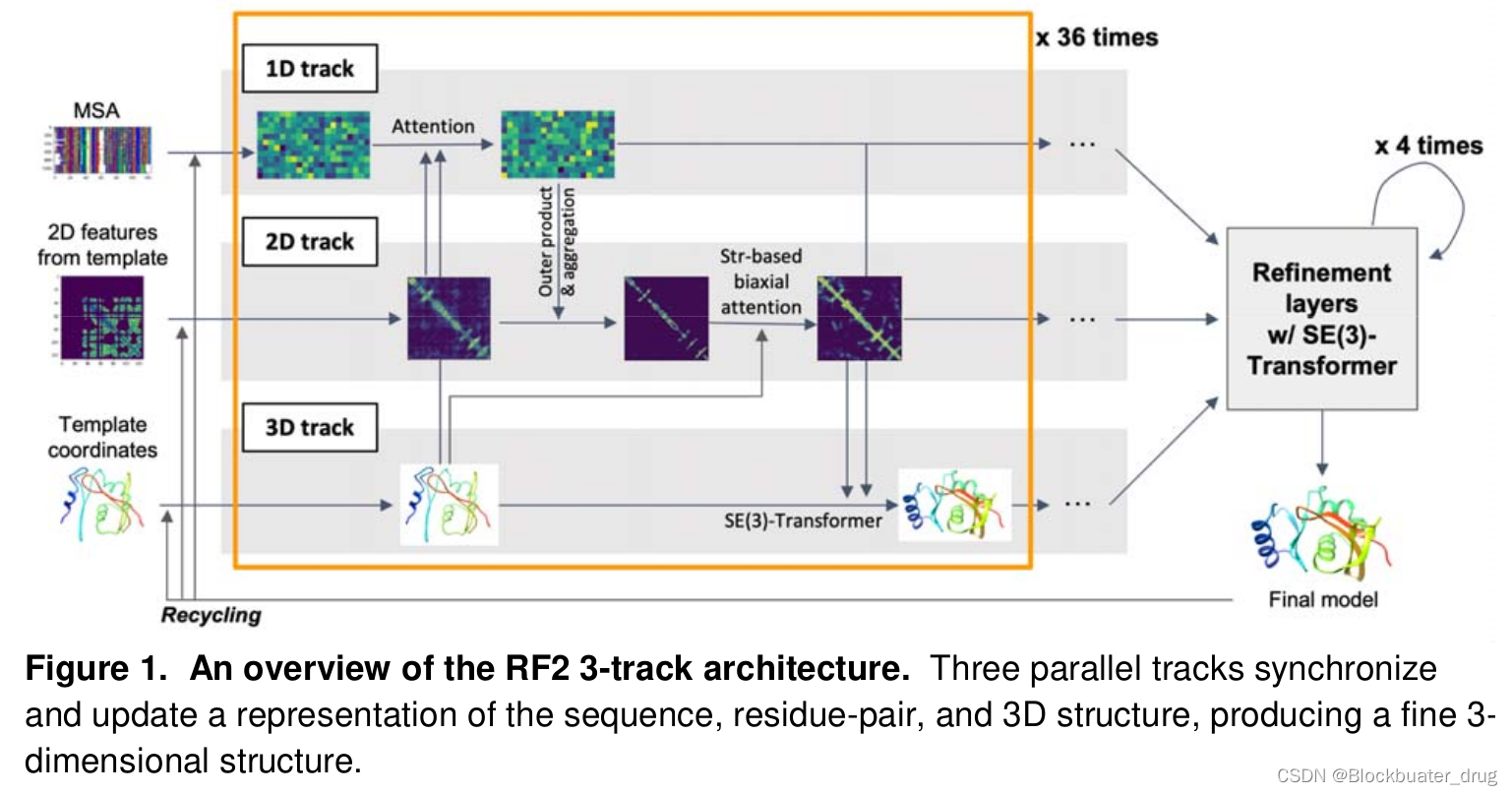
在整个网络上扩展了RoseTTAFold的原始三轨架构,结合了帧对齐点错误,训练过程中回收的概念,以及使用AlphaFold 2的数据集。作者还从AlphaFold 2中汲取了在更新配对特征时结构一致注意力的思想,但使用了计算效率更高的结构偏向注意力,而不是三角形注意力。由此产生的模型具有单体比的AlphaFold 2和复合物上比AlphaFold 2-multimer的准确性,对于大蛋白质和复合物具有更好的计算缩放。这种出色的性能是在没有AlphaFold 2的标志性特征,不变点注意力和三角形注意力的情况下实现的,这表明这些对于高精度预测并不重要。
就架构而言,RoseTTAFold和AF2之间的主要区别包括:(1)在RF基本块中包含第三个“3D结构”轨道,(2)在2D对轨道(RF)中使用双轴注意力与三角乘法和三角注意力(AF2),(3)在RF中使用SE3等变变换器3,而不是不变点注意力(IPA)在用于计算结构更新的AF2中,(4)网络的总体深度,在RF中具有8+4个2-轨道/3轨道层,而在AF2中具有4+48个完整msa/集群msa进化器层。
RoseTTAFold2是一个单一的模型,其精度相当于单体的AlphaFold2和复合物的AlphaFold2-multimer,在大于1000个残基的蛋白质和复合物上具有更好的计算耗时增长。
 RF2的工作原理(上图),看起来是综合了前辈们的技能。
RF2的工作原理(上图),看起来是综合了前辈们的技能。
 RF2在大于2000 AA的较大蛋白预测上更具有时间优势;在CASP14靶点的测试上,与AF2相当或更优。
RF2在大于2000 AA的较大蛋白预测上更具有时间优势;在CASP14靶点的测试上,与AF2相当或更优。
二、安装步骤
安装环境:Ubuntu 22.04, CUDA runtime版本11.8。
安装步骤:
Step 1. 创建conda环境
下载:
git clone https://github.com/uw-ipd/RoseTTAFold2.git
进入目录:
cd RoseTTAFold2
创建conda环境,名称RF2:
conda env create -f RF2-linux.yml
激活:
conda activate RF2
安装pandas:
conda install pandas
Step 2. 安装SE(3)-Transformer
安装 NVIDIA’s SE(3)-Transformer
保持RF2激活:
conda activate RF2
进入SE3T目录:
cd SE3Transformer
使用requirements安装依赖:
pip install --no-cache-dir -r requirements.txt
安装SE3T:
python setup.py install
遇到的问题2: 安装后pytorch是cpuonly版本,显示cuda不能work
解决方法: 运行 conda uninstall cpuonly,即可
Step 3. 下载预训练模型参数
下载预训练模型参数到network目录:
cd network
wget https://files.ipd.uw.edu/dimaio/RF2_apr23.tgz
tar xvfz RF2_apr23.tgz
cd ..
Step 4. 下载数据库
下载序列和结构数据库,有3和数据库需要下载:
uniref30 [46 GB]
wget -c http://wwwuser.gwdg.de/~compbiol/uniclust/2020_06/UniRef30_2020_06_hhsuite.tar.gz
mkdir -p UniRef30_2020_06
tar xfz UniRef30_2020_06_hhsuite.tar.gz -C ./UniRef30_2020_06
BFD [272 GB]
wget -c https://bfd.mmseqs.com/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gz
mkdir -p bfd
tar xfz bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gz -C ./bfd
PDB100 [65 GB]
包括 *_a3m.ffdata 和 *_a3m.ffindex文件
wget -c https://files.ipd.uw.edu/pub/RoseTTAFold/pdb100_2021Mar03.tar.gz
mkdir -p pdb100_2021Mar03
tar xfz pdb100_2021Mar03.tar.gz ./pdb100_2021Mar03
遇到的问题: 数据库下载问题, uniref30和BFD比较容易下载,也可以使用其他时间版本的,AlphaFold2下载的也可以; PDB100提供的链接国内暂时不可用。
解决方法: PDB100替代下载路径:https://wwwuser.gwdg.de/~compbiol/data/hhsuite/databases/hhsuite_dbs/pdb100_foldseek_230517.tar.gz;
解压,按运行run_RF2.sh的提示修改数据库文件名,或者添加软连接,即可。
三、使用步骤
运行方法
Usage: run_RF2.sh [-o|--outdir name] [-s|--symm symmgroup] [-p|--pair] [-h|--hhpred] input1.fasta ... inputN.fasta
Options:
-o|--outdir name: Write to this output directory
-s|--symm symmgroup (BETA): run with the specified spacegroup.
Understands Cn, Dn, T, I, O (with n an integer).
-p|--pair: If more than one chain is provided, pair MSAs based on taxonomy ID.
-h|--hhpred: Run hhpred to generate templates
从序列到结构预测实例:
1. 下载已解析结构等序列及pdb文件:
conda activate RF2
mkdir workfir && cd workdir
wget https://www.rcsb.org/fasta/entry/8I55
运行monomer 预测:
../run_RF2.sh 8I55 -o RF2_8I55
运行5 min。
结果在 RF2_8I55/models/中
2. plddt查看
在pymol中打开:
coloraf RF2-model_00_pred

3. PDB 8I55与AlphaFold2,ESMFold和RoseTTAFold2预测结果比较
AlphaFold2结果来自:AlphaFold2.3 conda版本详细安装与使用
ESMFold结果:ESMFold conda安装、使用及与AlphaFold的简单比较
下载pdb结构:
wget https://files.rcsb.org/download/8I55.pdb
RMSD比较: PDB 8i55(绿色)与RoseTTAFold2,AlphaFold2和ESMFold预测结果计算RMSD,分别为:0.419,0.387,0.373,整体均表现良好,ESMFold最优。
运行时间: 143个氨基酸残基,RoseTTAFold2,AlphaFold2和ESMFold运行时间分别为:~ 5 min,~ 25 min,< 1 min,ESMFold最快。



总结
本文介绍了蛋白结构预测开源程序RoseTTAFold2的安装及使用体验。
参考资料
- https://www.rcsb.org/structure/8I55
- https://www.biorxiv.org/content/10.1101/2023.05.24.542179v1
- https://github.com/masterwhook/RoseTTAFold2
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献6条内容
已为社区贡献6条内容





所有评论(0)