生物学重复好不好--看看样本相关性
文章目录引言数据计算相关系数映射相关系数到热图完整代码引言生物学实验中,常常需要设置重复,例如技术重复、生物学重复,以此确保不是个体的偶然变异对结果产生影响。以转录组数据为例,一般会设置3-5个生物学重复,如何确认生物学重复的效果好坏呢,方法有很多,可以计算两两样本之间的相关性,可以进行样本的PCA分析,或者绘制聚类热图,这里首先介绍样本相关性方法。我们将在R,使用Rstudio进行计算绘图。数据
引言
生物学实验中,常常需要设置重复,例如技术重复、生物学重复,以此确保不是个体的偶然变异对结果产生影响。以转录组数据为例,一般会设置3-5个生物学重复,如何确认生物学重复的效果好坏呢,方法有很多,可以计算两两样本之间的相关性,可以进行样本的PCA分析,或者绘制聚类热图,这里首先介绍样本相关性方法。
我们将在R,使用Rstudio进行计算绘图。
数据
转录组数据分析完成以后,我们会拿到基因表达矩阵,格式如下,行为基因,列为样本(也可以是行为样本,列为基因,在R中转置函数t()可以秒秒种搞定)。

计算相关系数
何谓相关?简单来说若你高我也高,你低我也低,或者你高我低都可以叫做相关。数理统计上通过计算相关系数来衡量,取值[-1, 1],负数表示负相关,正数表示正相关。在显著性的前提下,绝对值越大,相关性越强。绝对值为0, 无线性关系;绝对值为1表示完全线性相关。有Pearson, Spearman和 Kendall 三类相关系数,它们的特点是:
| 相关系数 | 适用变量类型 |假设条件|
|–|–|–|–|
| Pearson | 连续变量|1.服从正态分布,2.两个变量的标准差不为0 |
| Spearman|连续变量/等级数据| 成对等级相关数据即可|
|Kendall|有序分类变量| 成对等级相关数据即可|
可以看到除了Pearson相关系数对数据有严格要求外,其他两种的适用范围都比较广,当你不确定数据分布时,一般适用Spearman即可。
这里要计算样本之间的相关性,落实到代码中,其实就是分别计算数据列与列之间的相关系数。
## 设置工作路径
setwd('/Users/yut/Desktop/data')
fpkm <- read.table('control_case_fpkm.txt', header = T, row.names = 1) #header=T,第一行指定为列名,row.names=1指定第一列为行名
View(fpkm) #查看数据
## 计算样本之间的相关性
corr <- cor(fpkm, method = 'spearman') #cor函数计算两两样本(列与列)之间的相关系数
View(corr) #查看样本之间的相关系数
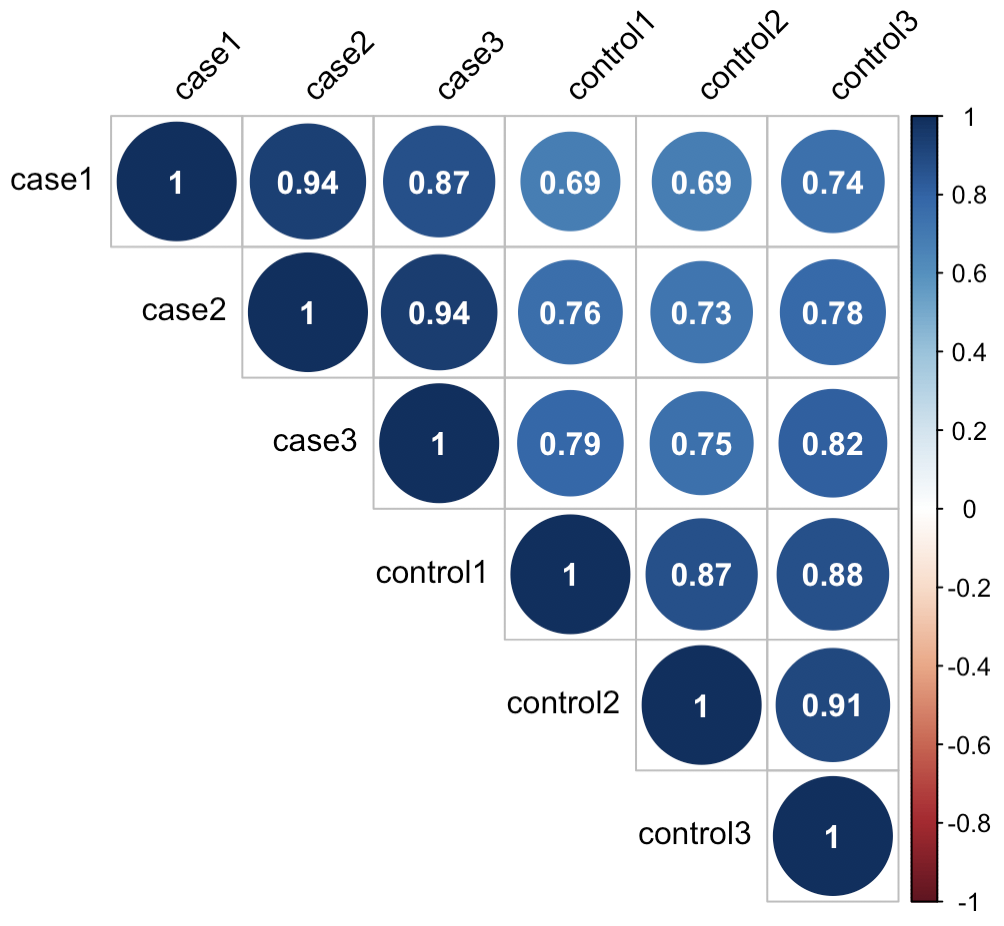
cor函数返回样本之间的相关系数矩阵,对角线为样本自身与自身的相关系数1,左下和右上半角是一样的

映射相关系数到热图
为了更直观的展示,使用corrplot将相关系数矩阵映射成热图
## 如果不存在corrplot就安装这个包
if (!requireNamespace('corrplot', quietly = TRUE))
install.packages('corrplot')
library('corrplot') #加载corrplot包用于绘制相关性矩阵热图
# corrplot并不能直接计算两两样本之间的相关系数,而需要通过R内置函数cor计算,corrplot真正做的是把cor计算得到的样本相关系数矩阵映射成不同的颜色和样式
# 映射成热图
corrplot(corr, type = 'upper', tl.col = 'black', order = 'hclust', tl.srt = 45, addCoef.col = 'white')
# type='upper':只显示右上角相关系数矩阵
# tl.col='black':字体颜色黑色
# order='hclust':使用层次聚类算法
# tl.srt = 45:x轴标签倾斜45度
# addCoef.col='white':添加相关系数数值,颜色白色

Encode计划建议皮尔逊相关系数的平方(R2)大于0.92(理想的取样和实验条件下)。具体的项目操作中,一般要求生物学重复样品间R2至少要大于0.8,否则需要对样品做出合适的解释,或者重新进行实验。一般来说:
0.8-1.0 极强相关
0.6-0.8 强相关
0.4-0.6 中等程度相关
0.2-0.4 弱相关
0.0-0.2 极弱相关或无相关
从上面的结果看,case组内和control组内的相关系数都较高,而组间的相关系数都较低,说明生物学重复效果好,可以继续后续分析。若组内某个样本与同组内其他样本的相关系数都较低,则可以考虑去除,或者加测样本进行重分析
corrplot输入
# corrplot并不能直接计算两两样本之间的相关系数,而需要通过R内置函数cor计算,corrplot真正做的是把cor计算得到的样本相关系数矩阵映射成不同的颜色和样式
d <- data.frame(
s1 = 1:3
, s2 = c(1, 0, 4)
, s3 = 5:7
)
corr <- cor(d)
> class(corr)
[1] "matrix" "array"
> corr
s1 s2 s3
s1 1.0000000 0.7205767 1.0000000
s2 0.7205767 1.0000000 0.7205767
s3 1.0000000 0.7205767 1.0000000
# 映射成热图
corrplot(corr, type = 'upper', tl.col = 'black', order = 'hclust', tl.srt = 45, addCoef.col = 'white')

完整代码
# <样本相关性>
## 1.如果不存在corrplot就安装这个包
if (!requireNamespace('corrplot', quietly = TRUE))
install.packages('corrplot')
library('corrplot') #加载corrplot包用于绘制相关性矩阵热图
# corrplot并不能直接计算两两样本之间的相关系数,而需要通过R内置函数cor计算,corrplot真正做的是把cor计算得到的样本相关系数矩阵映射成不同的颜色和样式
## 2.读入基因fpkm的表达矩阵
##设置工作路径
setwd('/Users/yut/Desktop/data')
fpkm <- read.table('control_case_fpkm.txt', header = T, row.names = 1) #header=T,第一行指定为列名,row.names=1指定第一列为行名
View(fpkm) #查看数据
##3.计算样本之间的相关性
corr <- cor(fpkm, method = 'spearman') #cor函数计算两两样本(列与列)之间的相关系数
View(corr) #查看样本之间的相关系数
# pdf('../sample_correlation.pdf', width = 8, height = 8) #打开绘图设备,保存为pdf文件
corrplot(corr, type = 'upper', tl.col = 'black', order = 'hclust', tl.srt = 45, addCoef.col = 'white')
# type='upper':只显示右上角相关系数矩阵
# tl.col='black':字体颜色黑色
# order='hclust':使用层次聚类算法
# tl.srt = 45:x轴标签倾斜45度
# addCoef.col='white':添加相关系数数值,颜色白色
# dev.off() #配合pdf()使用,关闭绘图设备

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献5条内容
已为社区贡献5条内容





所有评论(0)