转录组学习之序列比对(Hisat2)[学习笔记通俗易懂版]
Hisat2是一款短序列比对的工具,主要用于转录组数据的比对,是Hisat比对工具的升级版。Hisat2优化了索引建立的策略,采用了新的比对策略,使其与Bowtie/TopHat2等软件相比具有更高的敏感性和更快的运算速度。Hisat2支持剪切位点的识别和转录本的重构:Hisat2可以利用已知或发现的剪切位点信息进行剪切比对,提高比对率和准确性;同时,Hisat2可以结合StringTie等软件进
转录组学习之序列比对(hisat2)[学习笔记通俗易懂版]
data :2023.7.25
recorder :CYH-BI
特别注意:本文为我自己学习的学习记录,没有任何权威,只能仅供初学者提供思路与参考。
本文知乎地址:https://zhuanlan.zhihu.com/p/645765898
Hisat2介绍:
Hisat2是一款短序列比对的工具,主要用于转录组数据的比对,是Hisat比对工具的升级版。Hisat2优化了索引建立的策略,采用了新的比对策略,使其与Bowtie/TopHat2等软件相比具有更高的敏感性和更快的运算速度。Hisat2支持剪切位点的识别和转录本的重构:Hisat2可以利用已知或发现的剪切位点信息进行剪切比对,提高比对率和准确性;同时,Hisat2可以结合StringTie等软件进行转录本的重构和定量,提供更全面和精确的转录组信息。
原理 ( 文献 ):
HISAT: a fast spliced aligner with low memory requirements | Nature Methods
Download Hisat2 to RNA sequence Maching
方法一:官网下载安装包安装
一: 使用 wget 下载Hista2 安装包(该地址可能会变,仅供参考)
wget ftp://ftp.ccb.jhu.edu/pub/infphilo/hisat2/downloads/hisat2-2.1.0-Linux_x86_64.zip
解压(压缩包为 .zip文件,使用unzip解压)
unzip unzip /home/cyh/biosoft/hisat2-2.1.0-Linux_x86_64.zip
配置环境,将信息写入 ~/.bashrc或者 /etc/profile
vim ~/.bashrc #编辑该该文件
export PATH="/home/cyh/biosoft/hisat2-2.1.0: $PATH" #在末尾输入这行内容,路径是你自己的
source ~/.bashrc #重启文件,立即生效
方法二:使用conda安装
conda install -c bioconda hisat2
注意:使用该方法安装的前提你预先安装了miniconda3或anaconda
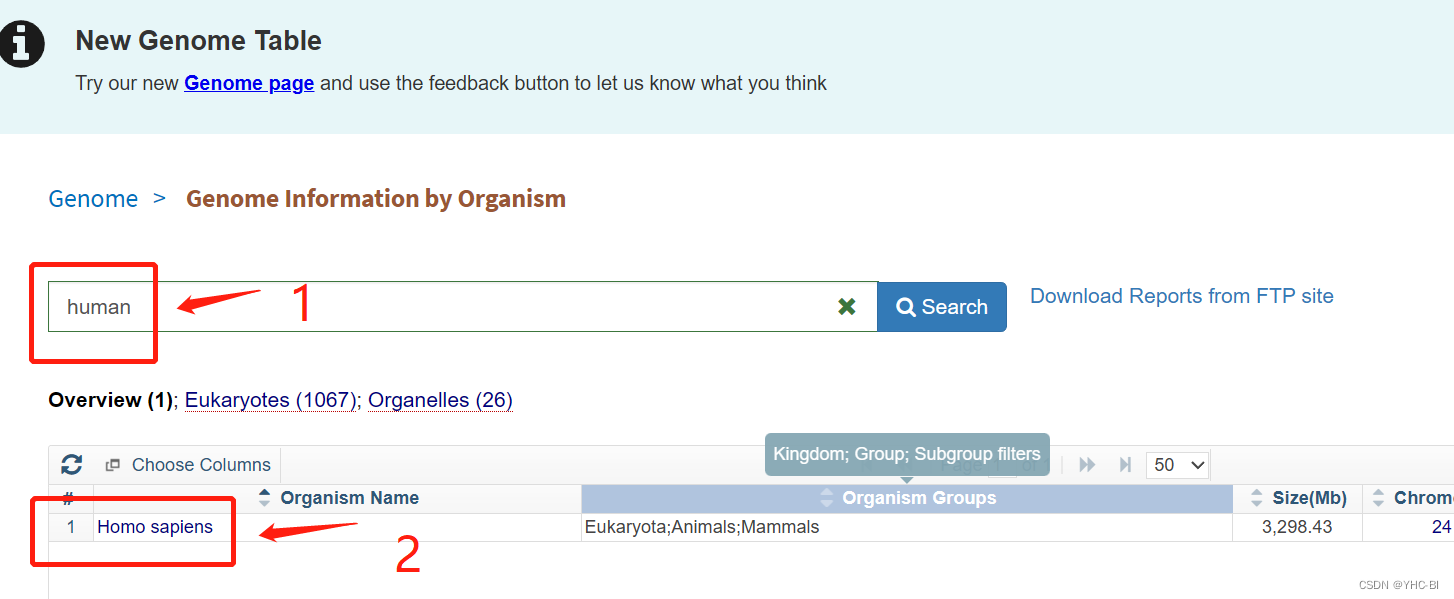
二:下载基因组文件(.fan格式)(以human为例)
下载地址:Genome List - Genome - NCBI (nih.gov) 如果是人类就在该页面搜索human

点击Homo sapiens

wget -c https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/001/405/GCF_000001405.40_GRCh38.p14/GCF_000001405.40_GRCh38.p14_genomic.fna.gz
-c 该参数防止断网或其他机器原因终端后导致下载失败,重新联网或排除故障后继续下载。
下载基因注释文件
wget -c https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/001/405/GCF_000001405.40_GRCh38.p14/GCF_000001405.40_GRCh38.p14_genomic.gff.gz
解压
gunzip GCF_000001405.40_GRCh38.p14_genomic.fna.gz
gunzip GCF_000001405.40_GRCh38.p14_genomic.gff.gz
三、手动建立索引
extract_exons.py /home/chenyh/ly_NT_RNAseq/reference_GRCh38/GCF_000001405.40_GRCh38.p14_genomic.gff > GRCh38.p14_genome.exon #(可选)
在构建HISAT2索引时,你需要准备基因组序列文件(通常是以.fa或.fasta为扩展名的文件)作为主要的输入数据。这个文件包含了基因组的DNA序列信息。
至于.gff文件,它通常用于注释基因组的功能元素,如基因、外显子、剪接位点等。在构建HISAT2索引时,如果你有.gff文件提供的注释信息,可以使用其他工具(如hisat2_extract_splice_sites.py和hisat2_extract_exons.py)将.gff文件转换为HISAT2所需的外显子(exon)和剪接位点(splice site)信息文件,然后将这些转换后的文件作为参数传递给hisat2-build命令。
因此,要构建HISAT2索引,你至少需要基因组序列文件作为输入数据,而.gff文件需要进行额外的处理和转换才能用于构建索引。(下面命令必选)
我脚本里的内容:
hisat2-build ./GCF_000001405.40_GRCh38.p14_genomic.fna GRCh38_index #index.sh
GRCh38_index 这是我使用的索引前缀
运行脚本:(将返回信息输入至index-log.txt)
nohup bash index.sh > 2>&1 &
四、序列比对
对于双端数据
hisat2 -p 4 --dta -x /home/cyh/Desktop/hugene_dir/GRCh38_index -1 /home/cyh/Desktop/fastq_dir/ly1_1.fq -2 /home/cyh/Desktop/fastq_dir/ly1_2.fq -S /home/cyh/Desktop/ly1_seq_mached.sam #脚本核心内容
GRCh38_index 是索引文件的前缀,索引文件共8个文件,格式:前缀.1.ht2、前缀.2.ht2…前缀.8.ht2,共8个文件,如下:其中GRCh38_index是前缀,比对的时候用到

nohup bash hisat2.sh > hisat2-log.txt 2>&1 & #运行时间长,后台运行代码
代码解释:
-p 4个核;
–dta :--dta参数用于生成RNA-Seq数据分析过程中的转录组装(transcriptome assembl;简称"TA")文件。当设置了--dta参数后,Hisat2在比对过程中会记录每个比对结果的详细信息,并将这些信息存储在转录组装文件中。
-1,第一个fq文件;-2 ,第二个fq文件;
-S ,输出文件为.sam格式;
对于单端数据
hisat2 -p 4 --dta -x /home/cyh/Desktop/hugene_dir/GRCh38_index -U input_file.fq -S /home/cyh/Desktop/ly1_seq_mached.sam
-U,表示单端数据
如需要查看hisat2的其他功能,请看使用手册或其他博主的博客
官网地址:Manual | HISAT2 (daehwankimlab.github.io)
推荐博主博文:
1、(4条消息) RNA-seq流程学习笔记(7)-使用Hisat2进行序列比对_垚垚爸爱学习的博客-CSDN博客
2、转录组分析 | 使用Hisat2进行序列比对-腾讯云开发者社区-腾讯云 (tencent.com)
题外内容 :
1、extract_splice_sites.py
extract_splice_sites.py genemo.gtf > genemo.ss
这是一个使用extract_splice_sites.py脚本从基因注释文件中提取剪接位点信息的命令。下面是各个参数的详细解释:
genemo.gtf :指定基因注释文件的路径和文件名,这里使用的是genemo基因组的基因注释文件。
genemo.ss :将提取的剪接位点信息输出到genemo.ss文件中。
综上,这个命令的作用是从genemo基因组的基因注释文件中提取剪接位点信息,并将结果输出到genemo.ss文件中。这个过程可以为后续的RNA测序数据分析提供剪接位点信息。
extract_splice_sites.py是一个用于从GTF或GFF3格式的基因注释文件中提取剪接位点信息的Python脚本。它可以生成一个包含所有剪接位点信息的文件,以供后续的RNA测序数据分析使用。使用extract_splice_sites.py时,需要指定输入的基因注释文件路径和文件名,以及输出剪接位点信息的文件路径和文件名。.ss文件是从基因注释文件中提取的剪接位点信息文件。这个文件包含了基因组上所有的剪接位点信息,包括剪接位点所在的染色体、位置、剪接类型等信息。这个文件通常用于RNA测序数据分析中的剪接分析,可以帮助识别不同的剪接事件并进行定量分析。
2、extract_exons.py
extract_exons.py genemo_data/genes/genemo.gtf >genemo.exon
这是一个使用extract_exons.py脚本从基因注释文件中提取外显子信息的命令。下面是各个参数的详细解释:
genemo_data/genes/genemo.gtf:指定基因注释文件的路径和文件名,这里使用的是genemo基因组的基因注释文件。
genemo.exon:将提取的外显子信息输出到genemo.exon文件中。
.exon文件是从基因注释文件中提取的外显子信息文件。这个文件包含了基因组上所有基因的外显子信息,包括外显子所在的染色体、位置、外显子编号、外显子长度等信息。这个文件通常用于RNA测序数据分析中的外显子定量分析,可以帮助识别外显子的边界和进行定量分析。
综上,这个命令的作用是从genemo基因组的基因注释文件中提取外显子信息,并将结果输出到genemo.exon文件中。这个过程可以为后续的RNA测序数据分析提供外显子信息。
3、hisat2-build
hisat2-build --ss genemo.ss --exon genemo.exon genemo_data/genome/genemo.fa genemo_tran
这是一个使用Hisat2软件构建基因组索引的命令。下面是各个参数的详细解释:
–ss genemo.ss:指定包含剪接位点信息的文件路径和文件名,这里使用的是从genemo基因组的基因注释文件中提取的剪接位点信息文件genemo.ss。
–exon genemo.exon:指定包含外显子信息的文件路径和文件名,这里使用的是从genemo基因组的基因注释文件中提取的外显子信息文件genemo.exon。
genemo_data/genome/genemo.fa:指定基因组序列文件的路径和文件名,这里使用的是genemo基因组的FASTA格式序列文件。
genemo_tran:指定输出的索引文件名,这里输出的索引文件将包含转录本信息,以便在RNA测序数据比对时考虑剪接事件和外显子信息。
这里使用的是从genemo基因组的基因注释文件中提取的外显子信息文件genemo.exon。
genemo_data/genome/genemo.fa:指定基因组序列文件的路径和文件名,这里使用的是genemo基因组的FASTA格式序列文件。
genemo_tran:指定输出的索引文件名,这里输出的索引文件将包含转录本信息,以便在RNA测序数据比对时考虑剪接事件和外显子信息。
综上,这个命令的作用是使用Hisat2软件构建基因组索引,将包含剪接位点信息和外显子信息的文件与基因组序列文件一起作为输入,构建包含转录本信息的索引文件genemo_tran。这个索引文件可以用于后续的RNA测序数据比对和分析。
到此,本文内容结束,这篇文章是经过了自己学习实践出来的,参考了很多资料,如若有大佬能指出错误,我将感激

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容





所有评论(0)