MRI脑影像分析——根据脑图谱获取感兴趣区域mask,以海马体与丘脑为例(matlab+nilearn+nibabel+REST1.8)
脑影像分析中,我们常常会针对性的对某些感兴趣区域进行分析,而对它们进行分析的前提是获取该区域的mask。感兴趣区域可以用以某些坐标为球心的球形区域定义,也可以用脑图谱上对应的某些脑区定义,其中,后者是较为常见的,也是我们今天要讨论的。脑图谱是一类特殊脑影像,它的每一个位置上不是信号值,而是脑区编号(标签),这也就意味着我们可以通过感兴趣区域的脑区编号,得到对应的感兴趣区域位置的集合,它也就是感兴趣

| 图源
脑影像分析中,我们常常会针对性的对某些感兴趣区域进行分析,而对它们进行分析的前提是获取该区域的mask。感兴趣区域可以用以某些坐标为球心的球形区域定义,也可以用脑图谱上对应的某些脑区定义,其中,后者是较为常见的,也是我们今天要讨论的。脑图谱是一类特殊脑影像,它的每一个位置上不是信号值,而是脑区编号(标签),这也就意味着我们可以通过感兴趣区域的脑区编号,得到对应的感兴趣区域位置的集合,它也就是感兴趣区域的mask。以下将以1mm和3mm的brainnetome图谱,并以海马体与丘脑为例生成mask,如果要生成其它mask,只需要修改编号序列即可。要用到的工具包括matlab、REST1.8、nilearn和nibabel等,从matlab到python,从GUI到代码脚本全方位覆盖,一起往下看吧。
copyright © 意疏:https://blog.csdn.net/sinat_35907936/article/details/118481241
获取海马体和丘脑的mask
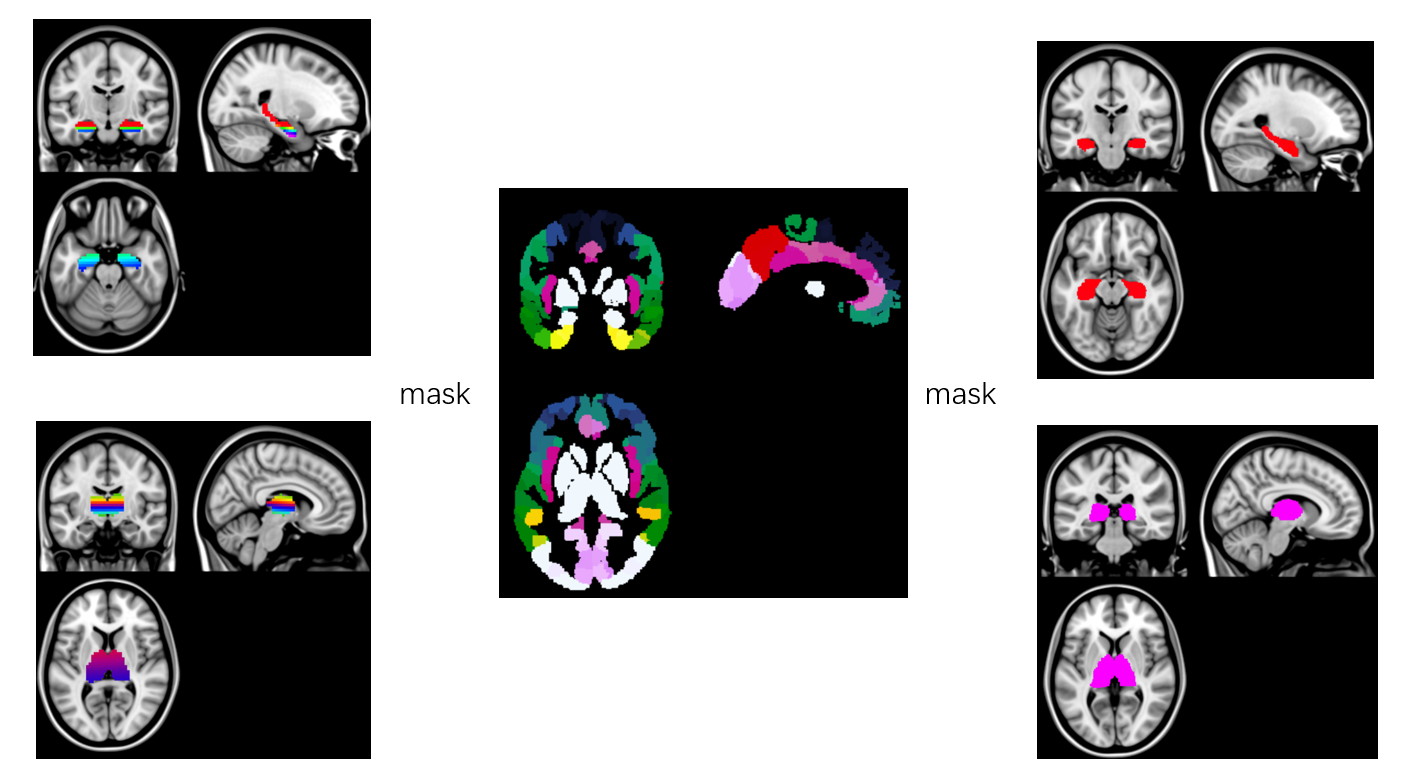
脑图谱是一类特殊脑影像,它的每一个位置上不是信号值,而是脑区编号(标签)。任何一个图谱都会有其对应的脑区编号(标签)表,brainnetone的编号表格可以在它的官网找到,这里就只截取了海马体与丘脑那部分。在图谱中,海马体与丘脑分别被分成了多个亚区,编号范围分别是215-218,231-246。

用mricron打开图谱图像,可以轻易地验证上述内容的正确性,即脑图谱每一个位置上不是信号值,而是脑区编号(标签),如下图。

REST1.8(静息态功能磁共振数据处理工具包 V1.8)是一个静息态脑影像分析matlab工具包,通过它,只需要点点点就可以获取到感兴趣区域的mask。基本步骤如下图所示,最关键的步骤是设置阈值范围,这也就是在通过选择脑区编号(标签)得到感兴趣区域,范围设置遵从matlab语法。另外在保存mask之前需要设置cluster大小,这对最终mask并没有什么影响,但是不能设置得太离谱,一般设置30 0 5,参照卢家峰老师的视频。

海马与丘脑mask:保存之后用mricron打开,以原来图谱为背景,设置颜色为grayscale,以mask为overlay,设置颜色为red,即可看到叠加之后的效果。

由上面的分析可知,只需要找到符合感兴趣区域对应的脑区编号范围内的所有位置就可以得到感兴趣区域的mask,这很容易实现,只需要解析NIFTI文件,再用阈值过滤数值即可。笔者在这篇文章里面详细的记录了各种解析NIFTI文件的方法,这里使用matlab自带的函数。只有0,1的mask方便用于提取该区域的全部信号,保留原始编号的mask则方便用于计算区域内各亚区的相关关系。
提取海马:215-218
ROI_label = 215:218;
area_name = 'hipp';
img = niftiread('BN_Atlas_246_1mm.nii');
info = niftiinfo('BN_Atlas_246_1mm.nii');
%创建一个空白mask
mask = uint8(zeros(size(img)));
for i = ROI_label
mask = mask + uint8(img==i);
end
% 区域内为1,区域外为0,一个编号
niftiwrite(mask,[area_name, '_uni_mask.nii'], info)
% 区域内为原来编号,区域外为0
niftiwrite(mask.*img,[area_name, '_sep_mask.nii'], info)

提取丘脑:231-246
ROI_label = [231:240,241:246];
area_name = 'tha';
img = niftiread('BN_Atlas_246_1mm.nii');
info = niftiinfo('BN_Atlas_246_1mm.nii');
%创建一个空白mask
mask = uint8(zeros(size(img)));
for i = ROI_label
mask = mask + uint8(img==i);
end
% 区域内为1,区域外为0,一个编号
niftiwrite(mask,[area_name, '_uni_mask.nii'], info)
% 区域内为原来编号,区域外为0
niftiwrite(mask.*img,[area_name, '_sep_mask.nii'], info)

nibabel是python平台上用于解析神经影像的工具,不仅可以解析nifti也可以解析gifti。这里用它来解析并封装nifti,以生成mask。mask类型与保持上面一致。
pip install nibabel
提取海马:215-218
import numpy as np
import nibabel as nib
area_name = 'hipp'
ROI_label = range(215,219)
img = nib.load('BN_Atlas_246_1mm.nii')
data = img.get_fdata()
mask = np.zeros(data.shape)
# 创建mask
for i in ROI_label:
mask[data==i] = 1
# 用原先的仿射矩阵与头包装mask成nifti
#区域内为1,区域外为0,一个编号
new_img = nib.Nifti1Image(mask,img.affine, header=img.header)
new_img.to_filename(area_name + '_uni_mask.nii')
# 区域内为原来编号,区域外为0
data = np.array(mask*data, dtype=np.uint8)
new_img = nib.Nifti1Image(data, img.affine, header=img.header)
new_img.to_filename(area_name + '_sep_mask.nii')
提取丘脑:231-246
import numpy as np
import nibabel as nib
area_name = 'tha'
ROI_label = list(range(231,240)) + list(range(240,247))
img = nib.load('BN_Atlas_246_1mm.nii')
data = img.get_fdata()
mask = np.zeros(data.shape)
# 创建mask
for i in ROI_label:
mask[data==i] = 1
# 用原先的仿射矩阵与头包装mask成nifti
#区域内为1,区域外为0,一个编号
new_img = nib.Nifti1Image(mask,img.affine, header=img.header)
new_img.to_filename(area_name + '_uni_mask.nii')
# 区域内为原来编号,区域外为0
data = np.array(mask*data, dtype=np.uint8)
new_img = nib.Nifti1Image(data, img.affine, header=img.header)
new_img.to_filename(area_name + '_sep_mask.nii')
# copyright © 意疏:https://blog.csdn.net/sinat_35907936/article/details/118481241
nilearn是在nibabel的基础上开发的,对nibabel做了一定程度的封装,并且集成了处理,统计分析和绘图等,是一个很完善的神经影像分析工具,这里也用它来提取一下丘脑和海马体的mask。与上面类似。
nilearn.image: 图像处理和重采样工具。
pip install nilearn
提取海马:215-218
from nilearn import image
import numpy as np
area_name = 'hipp'
ROI_label = range(215,219)
img = image.load_img('BN_Atlas_246_1mm.nii')
data = img.get_fdata()
# 创建mask
mask = np.zeros(data.shape)
for i in ROI_label:
mask[data==i] = 1
# 用原先的仿射矩阵与头包装mask成nifti
#区域内为1,区域外为0,一个编号
mask = mask.astype('uint8')
new_img = image.new_img_like(img, mask)
new_img.to_filename(area_name + '_uni_mask.nii')
# 区域内为原来编号,区域外为0
data = np.array(mask*data, dtype=np.uint8)
new_img = image.new_img_like(img, mask*data)
new_img.to_filename(area_name + '_sep_mask.nii')
提取丘脑:231-246
from nilearn import image
import numpy as np
area_name = 'tha'
ROI_label = list(range(231,240)) + list(range(240,247))
img = image.load_img('BN_Atlas_246_1mm.nii')
data = img.get_fdata()
# 创建mask
mask = np.zeros(data.shape)
for i in ROI_label:
mask[data==i] = 1
# 用原先的仿射矩阵与头包装mask成nifti
#区域内为1,区域外为0,一个编号
mask = mask.astype('uint8')
new_img = image.new_img_like(img, mask*data)
new_img.to_filename(area_name + '_uni_mask.nii')
# 区域内为原来编号,区域外为0
data = np.array(mask*data, dtype=np.uint8)
new_img = image.new_img_like(img, mask*data)
new_img.to_filename(area_name + '_sep_mask.nii')
copyright © 意疏:https://blog.csdn.net/sinat_35907936/article/details/118481241
补充——生成海马与丘脑区域内为每个体素编码的mask
为区域内每个体素编码的mask主要是方便用dpabi等工具算基于体素的功能连接,因为每个体素都有不同的编码,所以每个体素都会被当成一个ROI来对待,而不会被求平均。
提取海马:215-218
from nilearn import image
import numpy as np
area_name = 'hipp'
ROI_label = range(215,219)
img = image.load_img('BN_Atlas_246_3mm.nii')
data = img.get_fdata()
# 创建mask
mask = np.zeros(data.shape)
for i in ROI_label:
mask[data==i] = 1
# mask:区域内每个体素一个编号,区域外为0
idx_x, idx_y, idx_z = np.where(mask==1)
for j in range(len(idx_x)):
mask[idx_x[j]][idx_y[j]][idx_z[j]] = j
mask = np.array(mask, dtype=np.int16)
new_img = image.new_img_like(img, mask)
new_img.to_filename(area_name + '_all_mask.nii')
提取丘脑:231-246
from nilearn import image
import numpy as np
area_name = 'tha'
ROI_label = list(range(231,240)) + list(range(240,247))
img = image.load_img('BN_Atlas_246_3mm.nii')
data = img.get_fdata()
# 创建mask
mask = np.zeros(data.shape)
for i in ROI_label:
mask[data==i] = 1
# mask:区域内每个体素一个编号,区域外为0
idx_x, idx_y, idx_z = np.where(mask==1)
for j in range(len(idx_x)):
mask[idx_x[j]][idx_y[j]][idx_z[j]] = j
mask = np.array(mask, dtype=np.int16)
new_img = image.new_img_like(img, mask)
new_img.to_filename(area_name + '_all_mask.nii')

提取海马:215-218
ROI_label = 215:218;
area_name = 'hipp';
img = niftiread('BN_Atlas_246_3mm.nii');
info = niftiinfo('BN_Atlas_246_3mm.nii');
info.Datatype = 'uint16';%数字较大,需要修改原先的uint8到uint16
%创建mask
mask = uint16(zeros(size(img)));
for i = ROI_label
mask = mask + uint16(img==i);
end
idx = find(mask==1);
for i = 1:length(idx)
mask(idx(i))=i;
end
niftiwrite(mask,[area_name, '_all_mask.nii'], info)
提取丘脑:231-246
ROI_label = [231:240,241:246];
area_name = 'tha';
img = niftiread('BN_Atlas_246_3mm.nii');
info = niftiinfo('BN_Atlas_246_3mm.nii');
info.Datatype = 'uint16';
%创建mask
mask = uint16(zeros(size(img)));
for i = ROI_label
mask = mask + uint16(img==i);
end
idx = find(mask==1);
for i = 1:length(idx)
mask(idx(i))=i;
end
niftiwrite(mask,[area_name, '_all_mask.nii'], info)
注:其实只要能够解析nifti的工具都可以用来制作mask,上面只是举了几个例子,不过已经完全够用了。那么接下来就用这些生成的mask来提取感兴趣区域的信号吧,欢迎继续关注。
copyright © 意疏:https://blog.csdn.net/sinat_35907936/article/details/118481241
参考
https://blog.csdn.net/sinat_35907936/article/details/118862614

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献6条内容
已为社区贡献6条内容





所有评论(0)