(染色归一化)病理图像(HE或者WSI)图像标准化方法小介绍
虽说是CSDN,但因为处理的过程中要用到病理图片,所以这里简单的介绍一下病理图像的标准化。首先介绍一下我们的病理图像。病理图像就是我们高中时候用的玻片在扫描仪下拍摄的高分辨率图像。具体制作过程可以参见百度。https://wenku.baidu.com/view/60b5d0fe2af90242a995e58a.html病理图像可以帮助医生对病人做出诊断。通过病理图像,我们可以看到肿瘤细胞的...
更新
有朋友和我说Vahadane这个方法运行的时候有点慢。确实是不快,但对于很多更优的方法来说,已经是比较快的了。但确实比Macehko和Reinhard这种方法要慢不少。因为求非负矩阵求解的时候是迭代的。如果觉得时间太长,可以适当的减少迭代的次数。也可以多开几个进程,一次性把所有的图像都全部归一化,然后保存下来,以后就不用再做了。
更新
最近在arxiv上面有个用自监督做染色标准化的文章:
RestainNet: a self-supervised digital re-stainer for stain normalization
他在单个HE图像上面就可以训练,不需要任何的标注。训练完后,所有的图像都会标准化到训练所使用的WSI的颜色域上。
所使用的模型也是很简单。文中使用了GAN,但其实这个只要使用一个U-net就可以实现,使用GAN可能是为了使模型看起来复杂点。如果想使用深度学习做颜色标准化的,这个很简单也很实用,比较推荐。
更新:
给出一些比较常用的标准化方法。这些方法大多不是深度学习的方法,运行起来会比较快。深度学习的方法一般需要大量的训练数据,在新的数据集上泛化时效果可能也不太稳定。重新训练可能也找不到很好的训练数据。
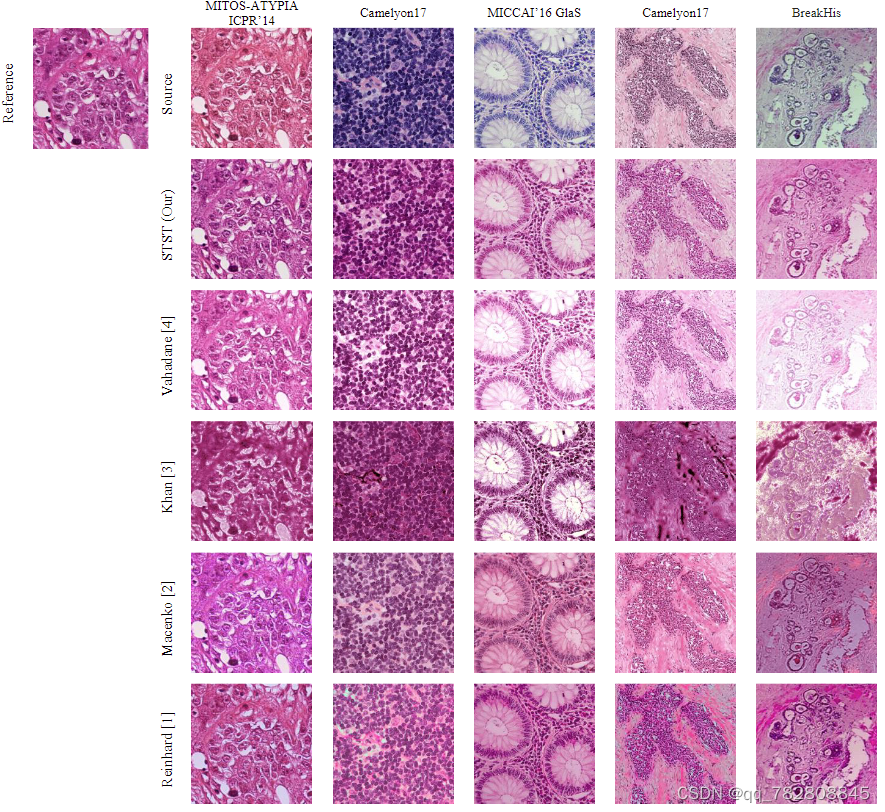
下图是来自文献[5]的截图,就是图中的STST。途中左上角的是目标风格,第一行source开始是未标化的图像。目的就是将source图像转换到reference风格上。所有的代码都可以在[5]中找到, 因为他公布了代码,并且提供了另外的方法的python实现。 我下面单独讲一下每一个算法。

STST: 这个方法是用c-gan做的,但我实际跑的时候效果很差,细胞核细胞质颜色很相近,分不开,可能因为他是对灰度图像染色,所以模型分不太开细胞核跟细胞质,不建议用。
Vahadane: 这个是我比较推荐的方法,也是本文最开始推荐的方法,代码就在文末。这个方法是用非负矩阵分解得到两个染料矩阵,然后将reference和source的进行配准,然后再合成新的图像。这个方法比较稳定,得到的图像颜色比较真实。实际的标化速度也可以接受。
Khan: 这个方法没怎么看,但冲效果里面看很差,不太建议用。
Macehko: 这个方法是将RGB转成OD,然后再用SVD分解得到两个垂直的颜色矩阵,再将这个颜色矩阵进行归一化,或者归一化到reference上。但我在实际应用的时候,得到的图像颜色不是很自然,会变得比较奇怪。一方面可能是SVD分解并没有非负分解这么好用,另一个可能是我reference图像选择不好。但速度上很快,效果一般。
Reinhard: 这个文章最开始并不是用在病理图像上的,它是用在自然图像上的,就是在Lab空间把两个图像的颜色进行统计学上的匹配。这个效果很差,但很快。差的原因很多,Lab颜色空间并不符合病理图像的光学特性,OD和HED更加符合他们的特性。其次,这个方法受输入图像的影响非常大,有些组织少的区域,为了得到相同的统计学分布,会将组织染色非常深,总之就很不智能。不推荐用。
如今还有很多很新的标化方法,但很多都没有公布代码,Vahadane这个方法是大多数研究常用的。新的方法可能标化的更好,但速度上可能不能在WSI上面使用。大家按照实际情况选择合适的算法。
[1] E. Reinhard, M. Ashikhmin, B. Gooch, and P. Shirley, “Color transfer between images,” IEEE Comput. Graph. Appl., vol. 21, no. 5, pp. 34–41, 2001.
[2] M. Macenko, M. Niethammer, J. S. Marron, D. Borland, J. T. Woosley, X. Guan, C. Schmitt, and N. E. Thomas, “A method for normalizing histology slides for quantitative analysis,” 2009 IEEE Int. Symp. Biomed. Imaging From Nano to Macro, pp. 1107–1110, 2009.
[3] A. M. Khan, N. Rajpoot, D. Treanor, and D. Magee, “A nonlinear mapping approach to stain normalization in digital histopathology images using image-specific color deconvolution,” IEEE Trans. Biomed. Eng., vol. 61, no. 6, pp. 1729–1738, 2014.
[4] A. Vahadane, T. Peng, A. Sethi, S. Albarqouni, L. Wang, M. Baust, K. Steiger, A. M. Schlitter, I. Esposito, and N. Navab, “Structure-Preserving Color Normalization and Sparse Stain Separation for Histological Images,” IEEE Trans. Med. Imaging, vol. 35, no. 8, pp. 1962–1971, 2016.
[5] Pix2pix-based stain-to-stain translation: A solution for robust stain normalization in histopathology images analysis.
更新:这里不少朋友找我要Structure-Preserving的代码。我这里引用一下原作者的[github],
但这个代码是用matlab写的,用起来比较麻烦,也有网友用python写了,我这里也引用一个,如果我侵权了,作者请联系我删掉。
这个方法包含了染色分离的求解,可以得到图像的染色通道,效果不错,而且速度也挺快。
正文
虽说是CSDN,但因为处理的过程中要用到病理图片,所以这里简单的介绍一下病理图像的标准化。
首先介绍一下我们的病理图像。
病理图像就是我们高中时候用的玻片在扫描仪下拍摄的高分辨率图像。具体制作过程可以参见百度。https://wenku.baidu.com/view/60b5d0fe2af90242a995e58a.html
病理图像可以帮助医生对病人做出诊断。通过病理图像,我们可以看到肿瘤细胞的具体情况。比如说有无浸润,有无淋巴结转移,分化程度如何。对肿瘤的诊断,预后,分级分期都是很有帮助的。
人工智能在病理图像的领域也有十分广泛的应用。可以用来做分割,分类,预后等。
随着人们研究的深入,病理图像的标准化变成了一个预处理的必要过程。那我们为什么要进行图像的标准化呢。
首先,病理图片的成像和很多因素有关系,制作过程,扫描的机器等都会对图像造成颜色的结构上面的不同。这种不同对人眼来说影响比较小,虽然颜色略有不同,但是还是可以分辨的。但是对于深度学习等算法来说就不太能适应这种不同。还有一个很大的原因是,也就是这种颜色和结构上的不同,使得不同机型, 不同机构甚至是不同操作员扫描出来的病理图片不能够放在一起用。
处理这种颜色差异的方法,就是颜色的标准化。叫做color normalization或者是stain normalization。原理就是将所有的图片都标准化到同一张模板图片的颜色分布上。
对于病理图片的标准化,从开始到现在大概有3种比较主流的方法。一个是灰度直方图的标准化,一个是颜色的标准化,一个是光谱的标准化。按照字面意思其实就是将所有图片都转化成模板图片灰度分布,将所有图片都转化成模板图片颜色(RGB)分布,将所有图片都转化成模板图片光谱(即染料的颜色空间,有多少种染料就有多少种颜色空间)分布。
前面两种的缺点是,灰度分布的方式要求两个图片的灰度分布都差不多,但往往图片的灰度分布差异还是挺大的,而且转化的过程中可能丢失很多结构和颜色的信息。颜色转换的方式可能会丢失结构信息,而且有监督的颜色转化算法计算量非常大。
最新的算法是一种保存结构和颜色标准化算法,属于光谱匹配的方式。主要的思想是利用稀疏矩阵分解的方法,将原图像通过最优化的方式分解成两个染料矩阵。他保存了图片的结构,而是标准化了颜色,并且计算量上面还是可以接受的。下面给出论文名字,论文中有matlab源码的下载链接:
Structure-Preserving Color Normalization and Sparse Stain Separation for Histological Images

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献3条内容
已为社区贡献3条内容






所有评论(0)