如何从NCBI上的Gene数据库批量下载基因序列数据
昨天先尝试了python脚本,为了保险起见,先编写了下载一个基因的压缩包数据,后续批量下载只需改一下ID的提供。但是比较有趣的是与官网直接手动下载相比,我编的程序下载的数据刚刚好缺了我最想要的gene.fna数据,也就是做了大半天无用功,真是drama哈哈哈。实现,但是通过Gene和Nucleotide数据库进行检索得到的结果不同,即Gene数据库检索结果不能直接得到fasta序列,它需要手动点击
目录
问题背景
在有基因名称的前提下,想要获得基因的fasta序列,就需要通过NCBI的Gene数据库检索。如果说基因的数量有很多个,想实现批量下载,可以使用NCBI的Batch Entrezhttps://www.ncbi.nlm.nih.gov/sites/batchentrez实现,但是通过Gene和Nucleotide数据库进行检索得到的结果不同,即Gene数据库检索结果不能直接得到fasta序列,它需要手动点击Download Datasets键获得基因数据压缩包,里面包含基因的fasta序列gene.fna文件。想要批量下载,就要使用编程来实现。

Batch Entrez检索(在Gene数据库提供基因ID文件,这里我提交了两个基因ID)

检索得到两个记录

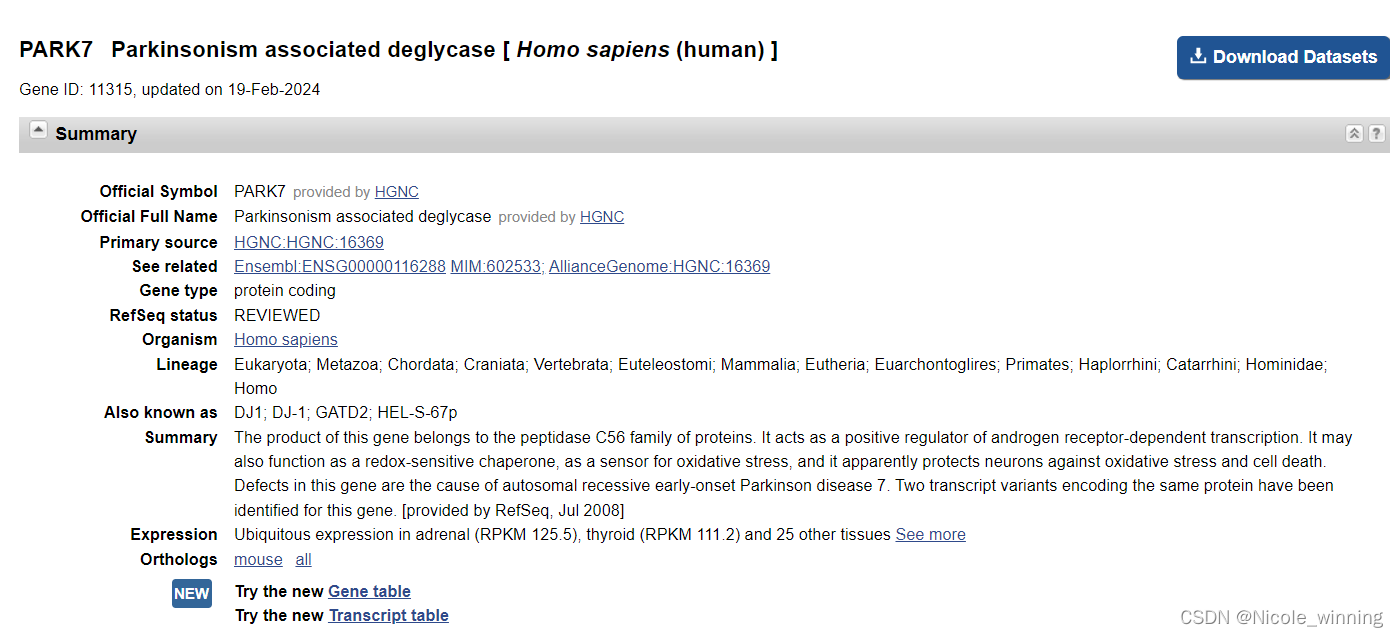
检索到的两个结果不能直接获得fasta序列
 需要点击Download Datasets获得基因fasta序列
需要点击Download Datasets获得基因fasta序列
解决问题
TBtools
TBtools是华南农业大学陈程杰老师开发的可用于生信分析的工具,也可以用于从NCBI上面下载数据以及对FASTA数据进行处理。想了解这个工具的可以访问一分钟入门并精通TBtools使用 · 语雀或者github上面Releases · CJ-Chen/TBtools-II · GitHub。
今天也尝试了使用TBtools来批量下载我想要的数据,但是这个软件也是用基因ID在nucleotide数据库进行检索,显然不是我想要的。但是这个工具还是比较好用的。

自己编程
既然网上的工具包括批量下载Batch Entrez和TBtools都不能解决,那么就得自食其力了。昨天先尝试了python脚本,为了保险起见,先编写了下载一个基因的压缩包数据,后续批量下载只需改一下ID的提供。但是比较有趣的是与官网直接手动下载相比,我编的程序下载的数据刚刚好缺了我最想要的gene.fna数据,也就是做了大半天无用功,真是drama哈哈哈。下面是我的初始代码:
import requests
# 基因ID
gene_id = "11315" # 检索基因ID,这个基因是PARK7
# 设置POST请求的URL
url = "https://api.ncbi.nlm.nih.gov/datasets/v2alpha/gene/download"
# 设置请求头
headers = {
"Content-Type": "application/json"
}
# 设置POST请求的数据
data = {
"gene_ids": [gene_id],
"file_types": ["FASTA_GENE"], # 设置为 "FASTA_GENE" 表示要下载基因序列的FASTA文件
"file_name": "PARK7_datasets.zip" # 设置文件名,可自定义
}
# 发送POST请求
response = requests.post(url, json=data,headers=headers)
# 检查响应状态码
if response.status_code == 200:
# 保存文件到本地
with open("PARK7_datasets.zip", "wb") as f:
f.write(response.content)
print("Gene Sequences (FASTA) 文件下载成功。")
else:
print("下载失败,状态码:", response.status_code)
给大家看一下脚本下载和官网下载的数据:

左为官网下载,右为脚本下载
虽然没有成功完全下载,但是大家可能比较好奇脚本是怎么写的,其实就是基于检索界面的网页源代码来写的,大家在检索界面右击“查看网页源代码”就行。

后来我想了一下,没有成功完全下载可能是因为API没有通过,所以去仔细看了一下NCBI官网是如何提供编程下载渠道的。
改进代码
通过官网"Documentation"部分,找到"Programming languages"查看帮助。Datasets Documentation



可以看到这里有示例代码,但需要先配置它这个包,我们先登服务器,在linux系统中来尝试一下。首先通过OpenAPI java libraries建立Build Python NCBI Datasets API v2alpha library,命令如下:
#!/usr/bin/env bash
OUTPUT_DIR="python_lib"
wget https://www.ncbi.nlm.nih.gov/datasets/docs/v2/openapi3/openapi3.docs.yaml
wget https://repo1.maven.org/maven2/org/openapitools/openapi-generator-cli/7.2.0/openapi-generator-cli-7.2.0.jar -O openapi-generator-cli.jar
java -jar openapi-generator-cli.jar generate \
-g python \
-i openapi3.docs.yaml \
--package-name "ncbi.datasets.openapi" \
--additional-properties=pythonAttrNoneIfUnset=true,projectName="ncbi-datasets-pylib"具体运行过程如下:
 配置完成后就可以运行修改后的代码了。在linux系统先安装一下我们配置的包,然后就可以运行py文件了。
配置完成后就可以运行修改后的代码了。在linux系统先安装一下我们配置的包,然后就可以运行py文件了。
pip install python_lib
python gene_fasta.py这里我会提供一个示例基因ID文件(包含20个基因及其ID),大家做的时候可以根据自己的数据提供基因ID文件。(文件链接:https://pan.baidu.com/s/1K5j7OSbkmnimiXGQYsl7rA?pwd=gy24 提取码:gy24 )具体代码如下:
import sys
import csv
import io
import os
from typing import List
from zipfile import ZipFile
from ncbi.datasets.openapi import ApiClient as DatasetsApiClient
from ncbi.datasets.openapi import ApiException as DatasetsApiException
from ncbi.datasets.openapi import GeneApi as DatasetsGeneApi
zipfile_name = "gene.zip" #自定义下载的压缩包名称
# 从CSV文件中读取基因ID并存储在列表中
gene_ids_from_csv = []
csv_file_path = "gi.csv" # 基因ID的CSV文件路径
with open(csv_file_path, newline='') as csvfile:
reader = csv.reader(csvfile)
for row in reader:
gene_id = int(row[1]) # 基因ID在CSV文件的第二列
gene_ids_from_csv.append(gene_id)
# 将基因ID列表转换为字符串格式,形如 "[1, 2, 3, ...]"
gene_ids_string = str(gene_ids_from_csv)
# 将字符串中的单引号替换为双引号,使其成为合法的Python列表表示形式
gene_ids_string = gene_ids_string.replace("'", '"')
# 将字符串转换为Python列表
gene_ids_list = eval(gene_ids_string)
with DatasetsApiClient() as api_client:
gene_ids: List[int] = gene_ids_list
gene_api = DatasetsGeneApi(api_client)
try:
gene_dataset_download = gene_api.download_gene_package_without_preload_content(
gene_ids,
include_annotation_type=["FASTA_GENE", "FASTA_PROTEIN"], #选择下载的数据格式
)
with open(zipfile_name, "wb") as f:
f.write(gene_dataset_download.data)
except DatasetsApiException as e:
sys.exit(f"Exception when calling GeneApi: {e}\n")
try:
dataset_zip = ZipFile(zipfile_name)
zinfo = dataset_zip.getinfo(os.path.join("ncbi_dataset/data", "protein.faa"))
with io.TextIOWrapper(dataset_zip.open(zinfo), encoding="utf8") as fh:
print(fh.read())
except KeyError as e:
logger.error("File %s not found in zipfile: %s", file_name, e)
这里只有20个基因,所以批量下载还是很快的,可以看一下下面的参考结果:


具体fna数据
问题补充
也许大家拿到手的是基因的名称文件,那么如何一键式查找到它们的基因ID呢,下面的代码可以帮助大家(根据自己情况更改存放基因名的文件try.csv):
import csv
from Bio import Entrez
def fetch_gene_ids(gene_names):
#Entrez.email = "...@..com" # 设置你的邮箱地址
gene_ids = {}
total_genes = len(gene_names)
for i, gene_name in enumerate(gene_names, 1):
print(f"Processing gene {i} of {total_genes}") #查看进程
search_term = f"{gene_name}[Gene Name] AND Homo[Organism]" #检索关键词,某个基因以及属于人类
handle = Entrez.esearch(db="gene", term=search_term) #利用Gene数据库检索
record = Entrez.read(handle)
handle.close()
if record["IdList"]:
gene_ids[gene_name] = record["IdList"][0]
else:
gene_ids[gene_name] = "N/A"
return gene_ids
def main():
gene_names = []
with open("try.csv", "r") as csvfile:
reader = csv.reader(csvfile) #提供一个存放基因名的文件
for row in reader:
gene_names.append(row[0])
gene_ids = fetch_gene_ids(gene_names)
with open("gene_ids.csv", "w", newline="") as csvfile: #输出基因ID
writer = csv.writer(csvfile)
writer.writerow(["gene_name", "gene_id"])
for gene_name, gene_id in gene_ids.items():
writer.writerow([gene_name, gene_id])
if __name__ == "__main__":
main()
其实这个一键式查询基因并找到基因ID还是比较耗时的,如果能直接得到基因ID的话,对后续获取fasta数据更便捷。

开放原子开发者工作坊旨在鼓励更多人参与开源活动,与志同道合的开发者们相互交流开发经验、分享开发心得、获取前沿技术趋势。工作坊有多种形式的开发者活动,如meetup、训练营等,主打技术交流,干货满满,真诚地邀请各位开发者共同参与!
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容





所有评论(0)