3D医学图像分割大模型 SegVol: Universal and Interactive Volumetric Medical Image Segmentation
我们提出了SegVol,一个交互式的通用体医学图像分割的基础模型。该模型是使用25个开源数据集开发和评估的。与最强大的传统体积分割方法nnU-net[9]不同,它会自动为每个数据集配置设置,SegVol的目的是将各种体积分割数据集统一到一个单一的架构中。在超过200个解剖目标产生准确的反应。此外,与传统方法相比,SegVol具有最先进或接近最先进的体积分割性能[5-10],特别是对于病变解剖目标。
SegVol: Universal and Interactive Volumetric Medical Image Segmentation
pub: 22 November, 2023 ArXiv
[ paper] [ code]
研究背景及动机#
背景:
1 体积图像分割通过准确提取器官、病变和组织等感兴趣的区域,在医学图像分析中起着至关重要的作用,在肿瘤监测、手术计划、疾病诊断和优化治疗等临床应用中有着广泛的应用。
2 公开可用的体积医学图像数据集通常由来自不同类别的少量掩码注释组成,由于模型训练的数据不足,也无法通过用户交互利用空间信息传统分割模型的泛化性较差,在分割肿瘤、囊肿等复杂结构时表现不佳。
3采用滑动窗口推理只包含局部信息且计算成本较高。
动机:
为了克服上述局限性,引入了一种通用的交互式体医学图像分割模型SegVol。
主要贡献
1 在96K的CT数据集上对模型进行预训练,并利用伪标签来解耦数据集和分割类别之间的虚假相关性。
2 将语言模型集成到分割模型中,并在25个数据集的200多个解剖类别上进行训练,从而实现文本提示分割。
3 采用协同策略协调语义提示和空间提示,实现高精度分割。
4设计一种**放大放大(zoom-out-zoom-in)**机制,显著降低计算成本,同时保持精确分割。
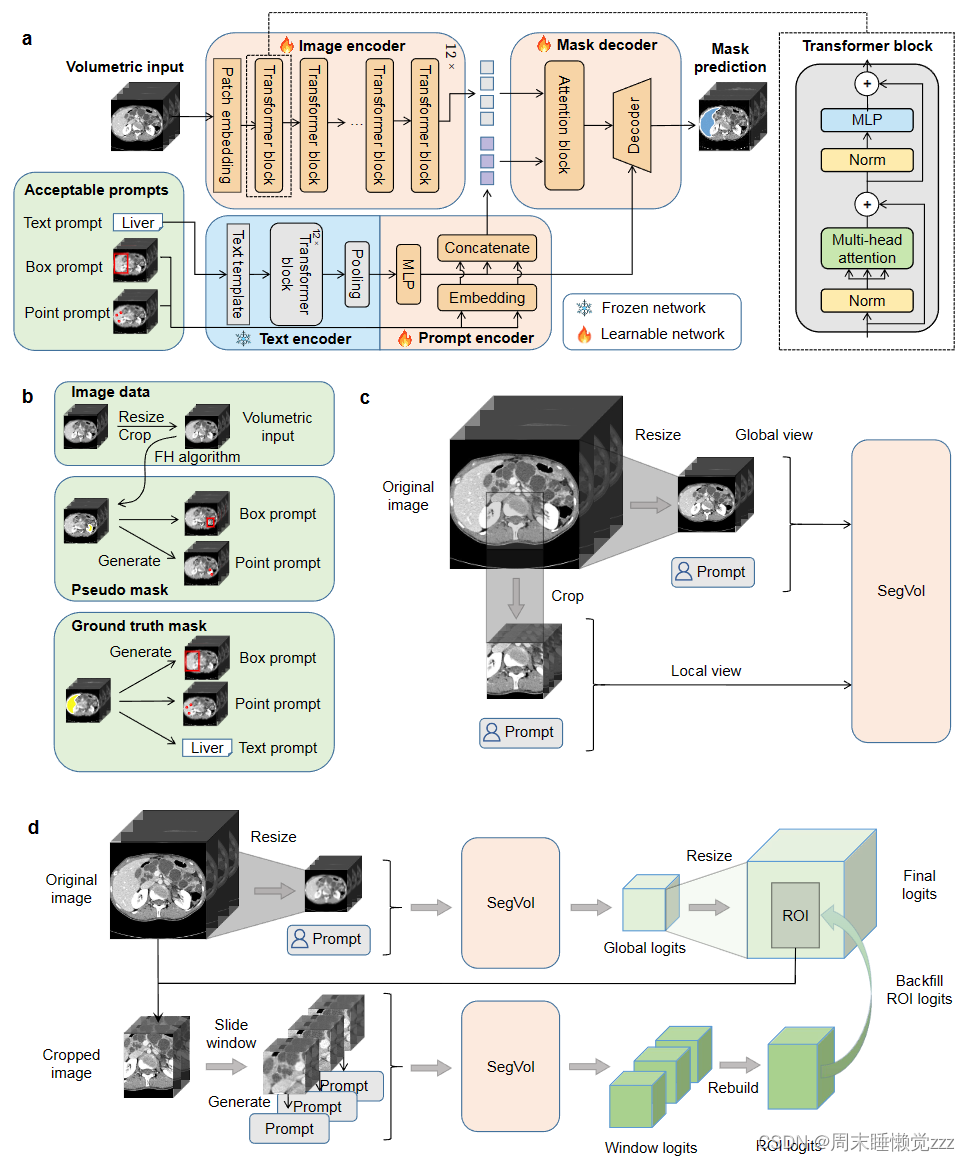
方法

b图为输入图像的变换和提示符的生成。
伪掩码生成与去噪:体分割数据集存在着部分标签的问题,大多数数据集只有少数分割目标的注释,例如几个器官。因此,深度模型可能会学习到数据集和分割目标之间的虚假相关性,从而在推理阶段产生较差的结果。为了消除这个问题,我们利用Felzenswalb-Huttenlocher (FH)[17]算法为每次CT扫描中的大多数物体生成伪掩码。无监督分割算法FH[17]基于相邻体素之间的梯度分离空间结构。但是,FH算法得到的伪掩码存在较大的噪声和大量的小掩码。该算法也可能导致分割不准确,例如某些连续结构的断开以及不同结构的错误连接。为了改进伪掩码,我们采用了以下策略:1)在适用的情况下用地面真值掩码替换伪掩码。2)我们过滤掉小于整体体积1‰的微小结构。3)每个伪掩码都经过膨胀和侵蚀操作精制而成。
放大放大机制:与二维幻灯片相比,体素数据具有非常大的体素数量,而分割目标相对较小。简单地对原始数据进行降采样会造成严重的信息丢失,从而降低性能。将大容量数据分解成小的立方体并分别处理每个立方体在计算上是昂贵的,而且还会造成信息丢失。为了降低计算成本,同时保留感兴趣区域(ROI)的细节,我们设计了由放大放大训练、放大放大推理组成的放大放大机制。
c图为放大放大训练:SegVol在全局和局部视图的数据上进行训练。
多视点的训练。为了适应不同大小的体积数据并实现放大-放大推理,我们构造了两类训练数据。一种是调整大尺寸CT的尺寸以适应模型的输入尺寸,获得放大视图的训练数据。另一种方法是根据模型的输入尺寸将原始大尺寸CT裁剪成立方体。这样,我们就得到了放大视图的训练数据。该过程如图1c所示。
d图为放大-放大推理:SegVol首先进行全局推理,然后对提取的ROI进行局部推理,以细化结果。
我们首先缩小并实现全局推理。给定一个大体积的图像,它被调整大小,然后输入到SegVol模型中。在获得全局预测后根据用户的提示定位感兴趣区域(ROI)并进行放大,即从原始尺寸的图像中裁剪出该区域。我们在裁剪区域上应用滑动窗口,实现更精确的局部推理。我们针对局部推理调整了输入提示,因为当放大时,用户输入的原始点框提示可能不适用于局部推理区域。具体来说,我们忽略局部区域外的正点或负点。与训练框提示生成类似,我们将局部区域的全局预测掩码作为伪掩码来生成局部框提示。最后用局部分割掩码填充全局分割掩码的ROI区域。放大放大机构同时实现了高效和精确的推理
为了建立SegVol:从25个开源医疗数据集中收集了6k(前面说的是96K?)个带有150k分割掩码注释的CT。此外,我们从网络上抓取了90k个未标记的CT数据,并获得了511k个由FH算法生成的伪体积掩码。
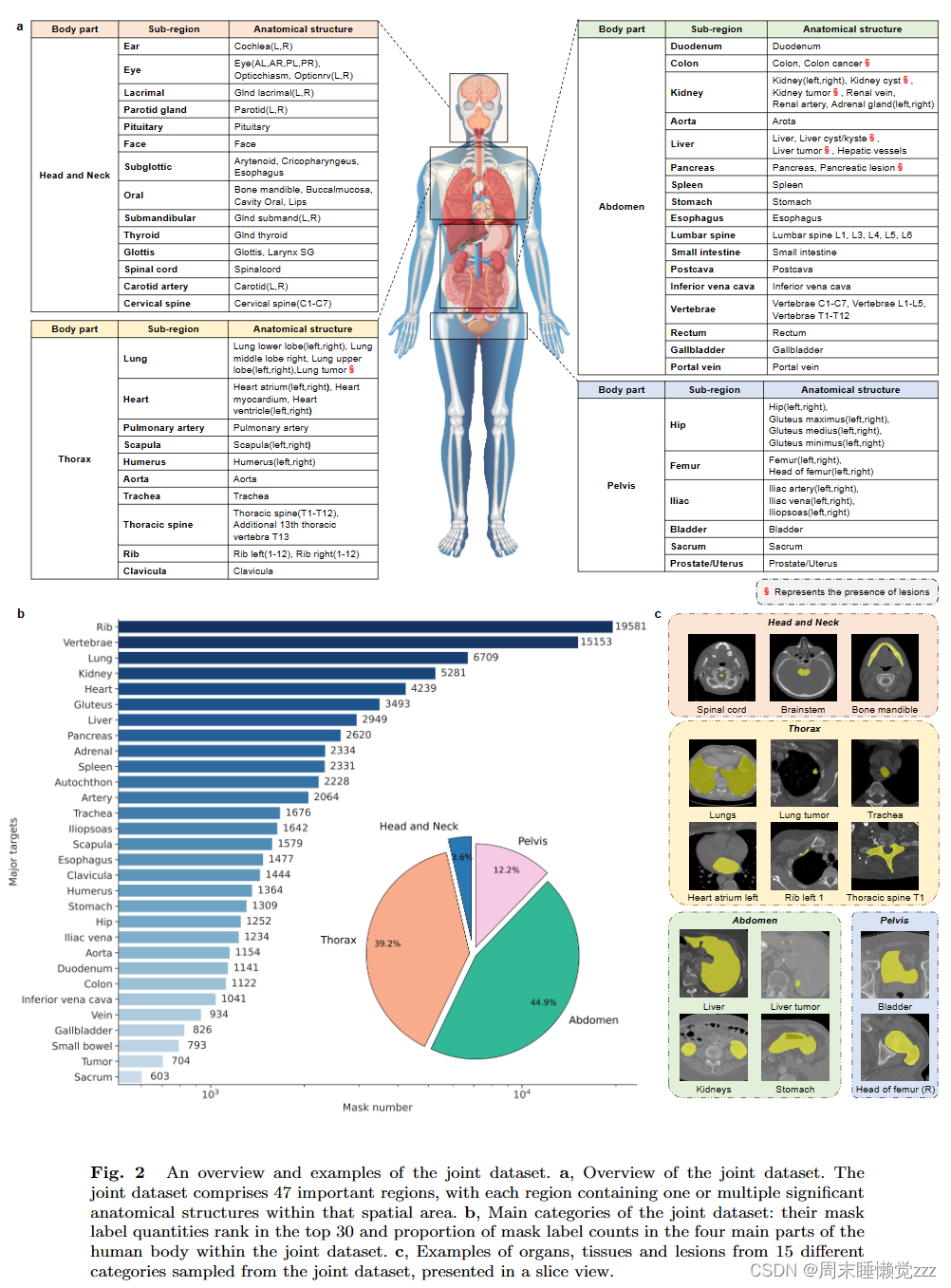
图2描述了数据集的组成:从医学开源数据集中收集了25个分割CT数据集,形成一个联合数据集,涵盖了CT图像分割的各种热点问题。图2a所示,收集到的关节数据集包括四个主要的人体区域:头颈部、胸腔、腹部和骨盆,包括47个重要区域的200多种器官、组织和病变类型。共有5772个CT参与了联合数据集的训练和测试,总共有149199个带语义的体积掩码标签。图2c中显示了四个主要区域的二维切片形式,为了增强SegVol的空间分割能力,使用FH算法为这些实例生成510k的伪体积掩码标签来填充未注释的区域。图2 b显示了关节数据集的前30个主要类别和联合数据集中人体四个主要部位的掩码标签分布情况。
在训练和测试过程中,联合数据集的每个子集被分为80%的训练数据和20%的测试数据。
使用Dice Similarity Coefficient (Dice score)作为度量来评价模型,定义为DSC = 2|X∩Y | |X|+|Y |。
数据处理和归一化
体积医学图像分割的主要挑战之一是缺乏大规模公开可用的体积医学数据,特别是带注释的分割ct。我们尽最大努力,收集了25个开源分割CT数据集,包括CHAOS[20-22]、HaN-Seg[23]、AMOS22[24]、腹部扫描[25]、KiTS23[26]、KiPA22[27-30]、KiTS19[31]、BTCV[18]、胰脏扫描[14,32,33]、3D-IRCADb[34]、腹部扫描[19,35]、TotalSegmentator[36]、CT- org[11 - 14]、VerSe19、VerSe20[37-39]、SLIVER07[40]、QUBIQ[41]、6个MSD数据集[19]、LUNA16[15]、WORD[42]。这些ct来自不同的医疗机构,由不同的机器捕获,具有不同的参数设置和扫描区域。这些因素导致了数据分布的显著差异,给数据处理带来了巨大的挑战。为了标准化这些数据集,我们对每个CT扫描执行以下转换:我们首先根据每个体素的平均值计算一个阈值,计算前景体素的99.95和0.05百分位数,并将它们作为裁剪原始体素的上限和下限。我们使用均值和标准差进一步规范化前景体素。高于这个阈值的体素被认为是前景。
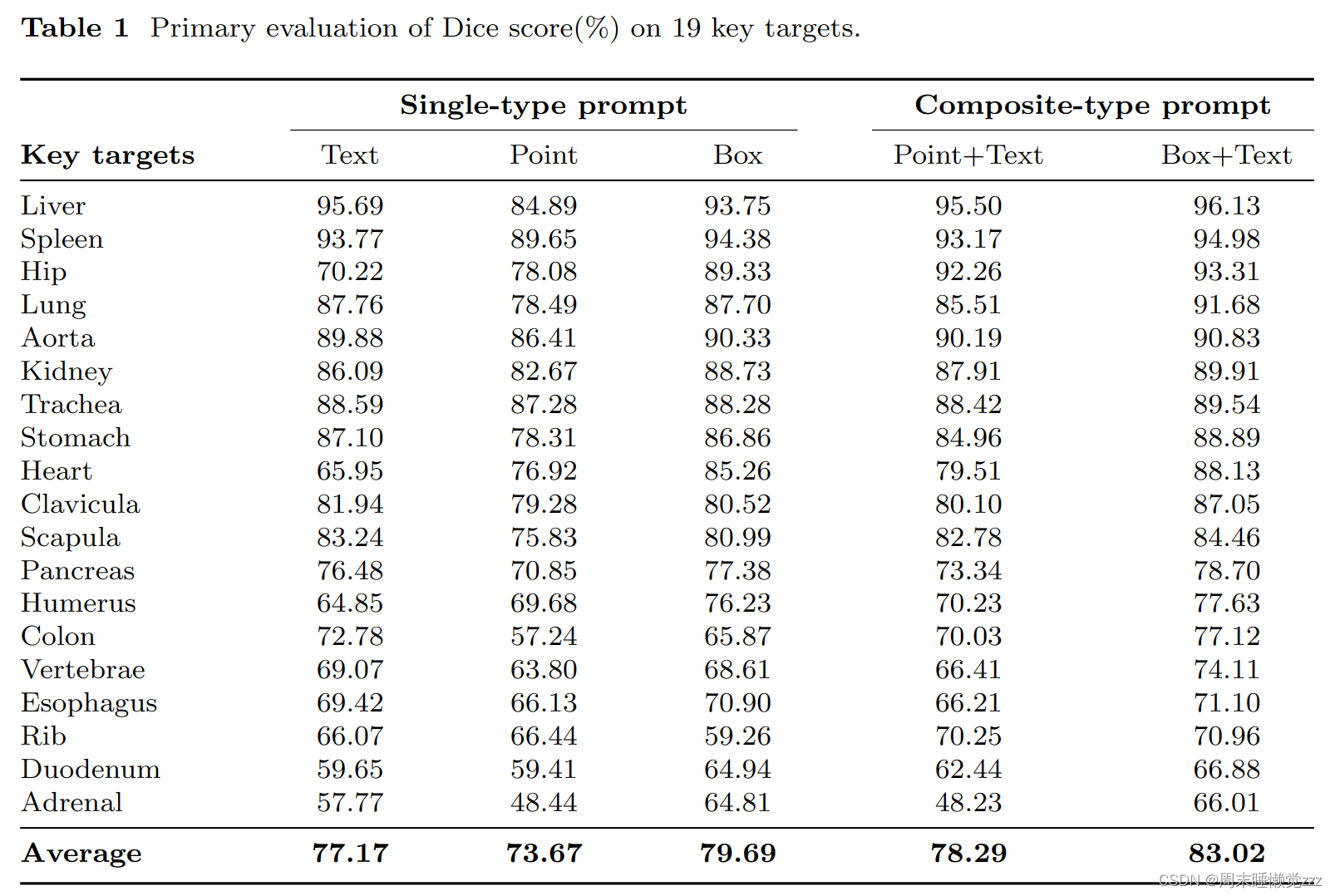
在提示学习的支持下,SegVol能够细分200多个类别。我们选择了19个重要的解剖目标来展示其强大的分割能力,如表1所示。SegVol展示了其对这些重要目标进行分割的卓越能力。肝脏的Dice得分高达96.13%,而19个主要目标的平均得分为83.02%,令人印象深刻。其强大的通用分割能力来自于具有语义和空间视角的复合型提示。一方面,空间提示可以让模型理解所涉及的具体空间和位置。由表1可知,在各种器官分割结果的平均水平上,“框+文本”提示的Dice分数比文本提示提高了5.85%。另一方面,语义提示澄清了对解剖结构的参考,消除了多个似是而非的推论。这反映在表1中,“点+文字”提示的平均骰子分数比单独使用点数提示高4.62%。空间提示和语义提示相互支持,最终赋予该模型强大的分割能力。
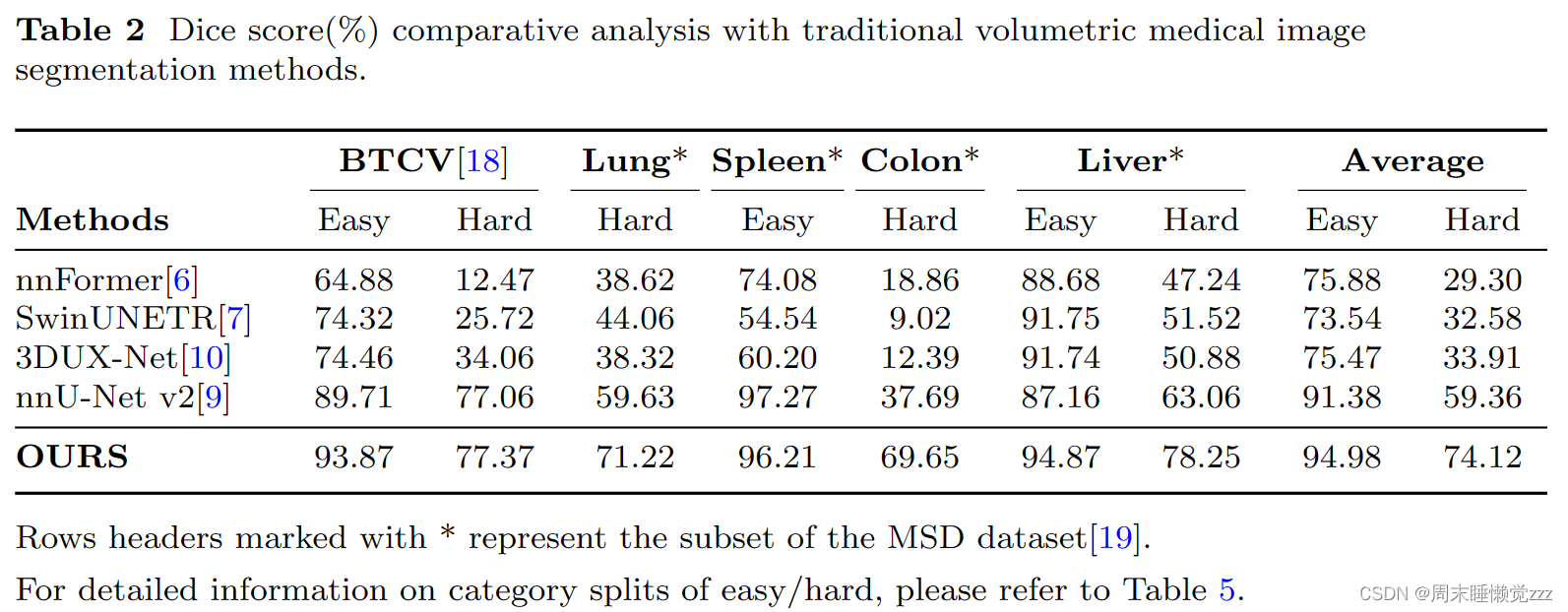
分割模型主要分为两种架构,基于cnn的模型和基于transformer的模型。我们对具有代表性的基于cnn的模型,如3DUX-Net(ICLR)[10]和nnU-Net(Nature methods)[9],以及具有代表性的基于变压器的模型,如nnFormer[6]和SwinUNETR[7]进行了对比实验。为了评估模型在器官、组织和病变分割方面的能力,我们在关注器官分割问题的BTCV[18]和msd -脾[19]数据集,以及关注病变分割问题的msd -肺、msd -结肠和msd -肝数据集上进行了对比实验。由于模型对“MegaStructures”和“MicroStructures”解剖目标的分割响应存在显著差异,为了更好地理解不同模型的性能差异,我们将分类分为易组和难组。有关类别划分的详细信息,请参见表5。
实验中,原始的开源训练集被设计成80%用于训练,20%用于测试。实验结果总结如表2所示。对于从数十到数百个病例的经典体医学图像数据集,SegVol在25个数据集上联合训练,显著优于在单个数据集上训练的传统分割模型。从表2可以看出,SegVol在肝、肾、脾等简单类别上优于传统模型,Dice得分达到94.98%。这主要是由于它从其他数据集的相同类别中学到了更多的知识。更重要的是,我们的方法在肝肿瘤、肺肿瘤和肾上腺等硬类别的分割中保持领先地位。SegVol对硬类的平均Dice得分绝对值比排名第二的nnU-net[9]高14.76%,对硬样本进行了精确分割。原因是SegVol可以通过空间和语义提示获得先验信息,从而增强对硬样本的理解,显著改善分割结果。
我们分析了使SegVol显著优于传统模型的主要因素有三个:1)固定类集的有限情况限制了传统模型的性能。相反,SegVol具有更广泛的学习范围,因为它结合了25个数据集进行训练。这使得它不仅可以从不同数据集的相同类别中收集知识,还可以从自然语言嵌入空间内固有相关的类别中收集知识。例如,SegVol可以从“左肾”和“肾”类别中学习,因为它们的自然语言相关性。这种从更广泛、更多样化的数据中学习的能力比传统模型更有优势,使其能够理解传统模型可能错过的分割目标的内在相关性。2)传统模型仅仅依靠整数代码来发现语义信息,而SegVol采用了更全面的快速学习方法。它不仅利用语义提示来理解目标,还利用空间提示来获得对目标的进一步空间认知。这种交互式分割模式使SegVol能够取得明显更好的结果,特别是在硬案例的精确分割中。3)对大规模未标记数据的预训练使SegVol能够学习更广义的特征表示。该过程显著增强了其对下游任务的适应性和鲁棒性。
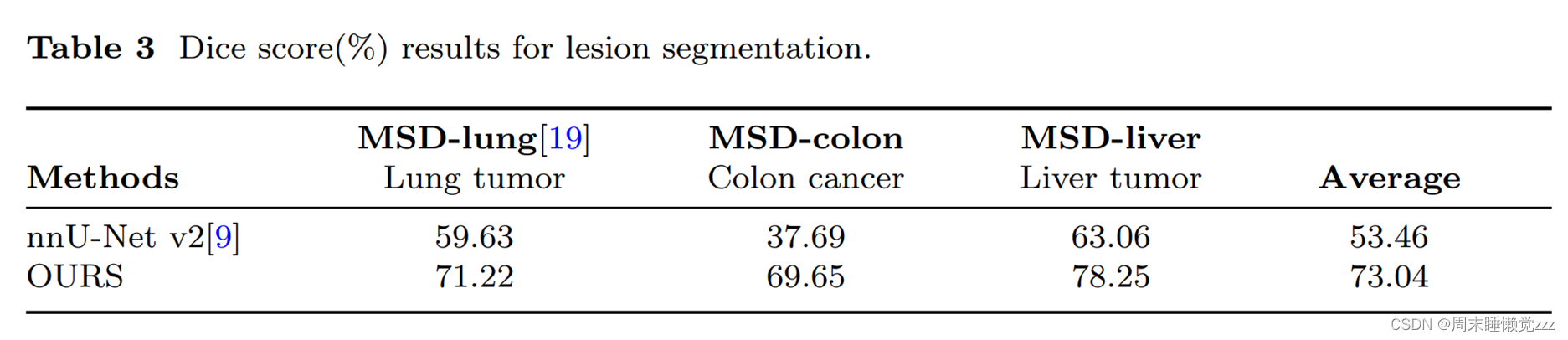
使用nnU-net[9]作为基线模型,它在传统的体积医学图像分割模型中表现出最强的分割能力。如表3所示,SegVol分割这些具有挑战性的病变病例的能力明显优于nnU-net。在这三个病变数据集中,SegVol的Dice评分绝对值超过nnU-net 19.58%,这代表了在复杂体积病例分割方面的重大进步。
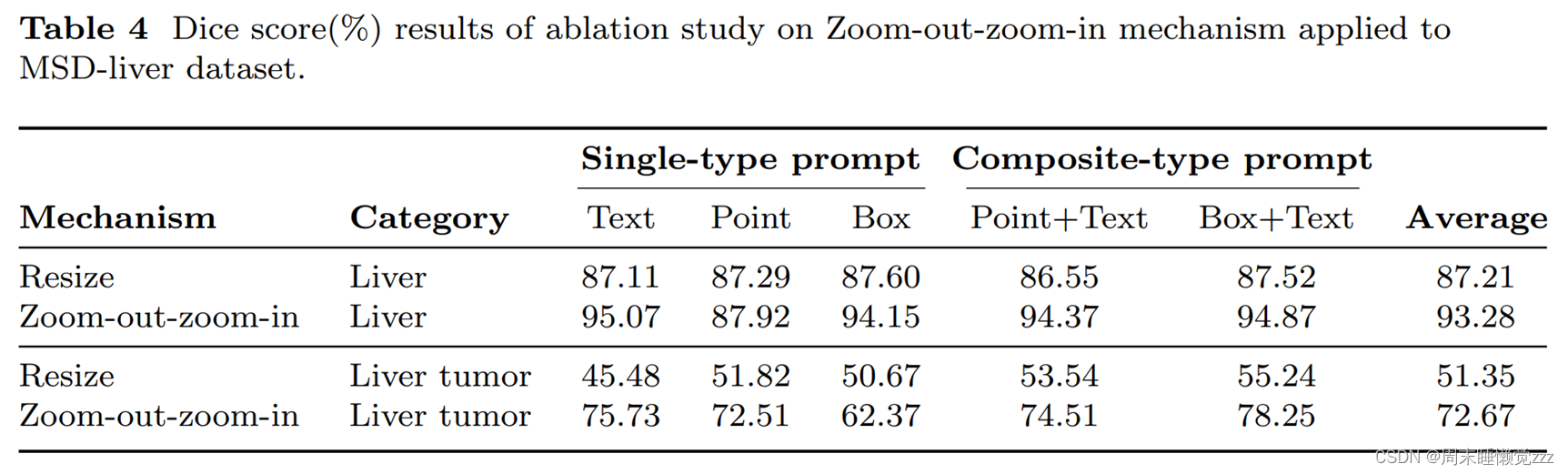
在msd -肝脏数据集上进行了消融研究[19],以评估Zoom-out-zoom-in机制的贡献。msd -肝脏数据集包括肝脏和肝脏肿瘤的类别,允许研究Zoomout-zoom-in机制对“MegaStructures”和“MicroStructures”目标的影响。如表4所示,将Zoom-out-zoom-in机制应用于SegVol模型导致肝脏类别的Dice评分提高了6.07%。这种改善在肝脏肿瘤类别中更为明显,其中Zoom-out-zoom-in机制将SegVol的Dice得分提高了21.32%。有趣的是,应用Zoom-out-zoom-in机制,在肝脏靶点提示设置内仅略有改善。我们认为,这可能归因于全球一级的点提示相对稀缺,当焦点缩小到局部区域时,这一点变得更加明显,从而限制了增加的潜力。

由于模型对“MegaStructures”和“MicroStructures”解剖目标的分割响应存在显著差异,为了更好地理解不同模型的性能差异,我们将分类分为易组和难组。有关类别划分的详细信息,请参见表5。

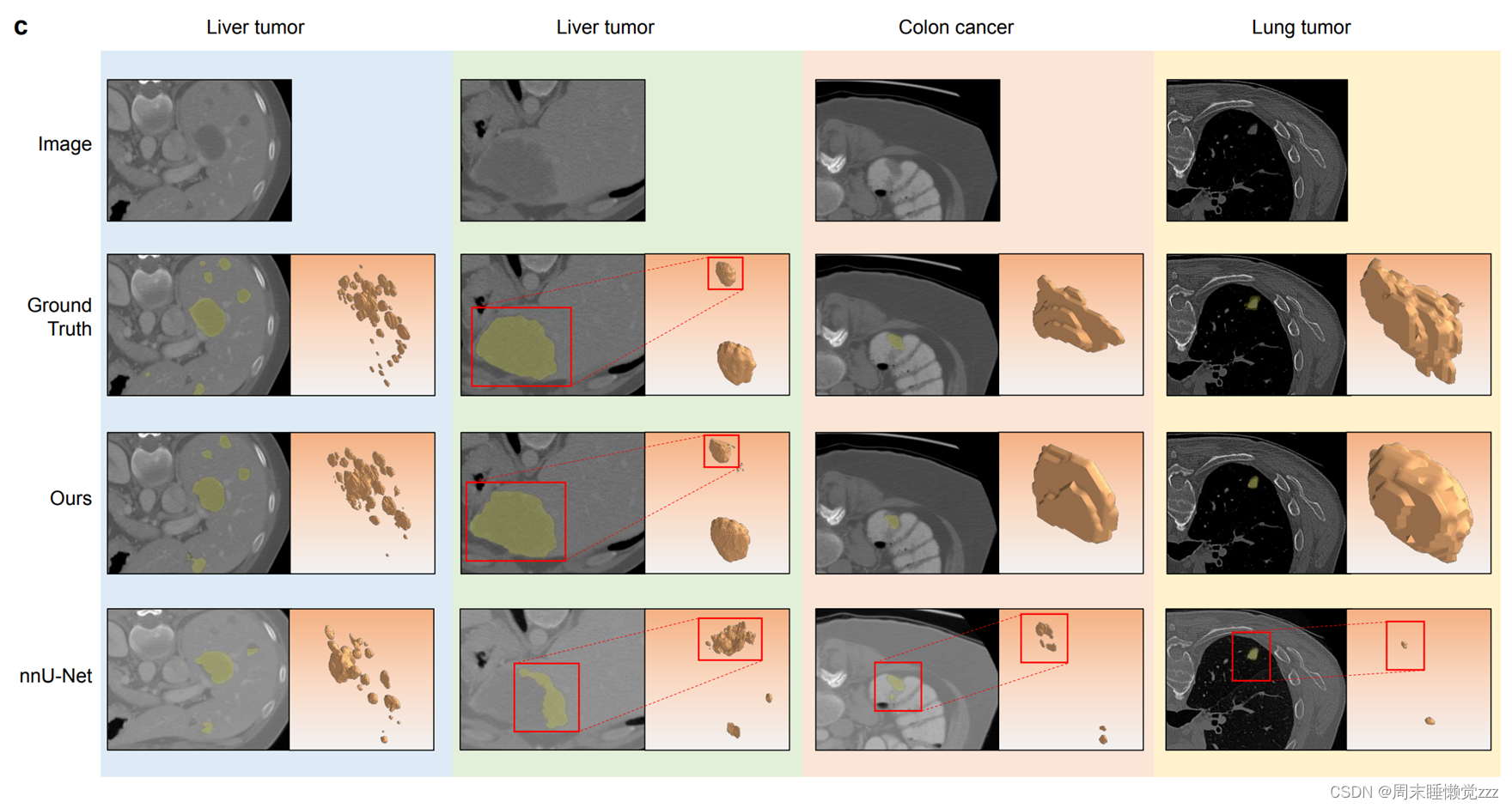
图3数据量尺度与病灶分割实验结果分析。a,在不同数量的数据集中,计算机断层扫描(CT)量和相应的地面真值掩模量的柱状图表示。b,描述在不同数据集上训练的模型的Dice分数的线形图。c,病灶分割结果的可视化表示。
数据规模是基础模型构建的关键因素之一。我们进行了消融研究,以研究图像和掩模的数量对模型性能的影响。以包含13个重要器官的BTCV数据集[18]为锚点,分别在1、2和8个数据集上训练了500个epoch的模型,以及在25个数据集上训练的最终模型进行评估。详细的结果如图3 a和b所示。作为轻量级模型,当只使用一个数据集时,性能不是最优的。然而,随着数据量的增加,模型的Dice得分显著增加,特别是在文本提示设置中,它严重依赖于带有语义信息的ground truth mask的数量。
图3c给出了一系列示例,说明了nnUnet和我们的方法的病变分割性能。这些例子包括肝肿瘤、结肠癌和肺肿瘤。可视化结果显示,与nnU-net产生的结果相比,SegVol重建的这些病变解剖结构更接近于ground truth。
总结
我们提出了SegVol,一个交互式的通用体医学图像分割的基础模型。该模型是使用25个开源数据集开发和评估的。与最强大的传统体积分割方法nnU-net[9]不同,它会自动为每个数据集配置设置,SegVol的目的是将各种体积分割数据集统一到一个单一的架构中。
在超过200个解剖目标产生准确的反应。此外,与传统方法相比,SegVol具有最先进或接近最先进的体积分割性能[5-10],特别是对于病变解剖目标。尽管具有通用性和精确性,但与其他体积分割方法相比,SegVol保持了轻量级架构。我们已经使SegVol成为一个开源的基础模型,很容易适用于广泛的医学图像表示和分析领域。这确保了它可以很容易地被研究人员和从业人员集成和利用。
传统的体积图像分割方法[5-10],由于其表述依赖 整数编码(integer codes)来表示语义信息,无法对不同解剖类别之间的相互关系进行建模。为了解决这个问题,我们使用提示学习技术来驱动SegVol。语义提示用于在自然语言嵌入空间中建模各种解剖类别,并使用空间提示符专门指代器官、组织和病变的空间结构。接受提示的能力使SegVol成为体积医学图像分割的通用和精确模型。为了减少与体积图像相关的计算成本,我们提出了一种放大放大机制。它允许用户在全局视图中简单地提示模型,同时在原始分辨率中 获得快速响应。这种创新的方法确保了医学图像分析的效率和精度。
虽然SegVol能够理解由句子组成的语义提示,但它与涉及复杂语义信息和逻辑关系的引用表达式分割之间仍然存在差距。引用表达式切分模型的建立需要更多具有逻辑关系的相关数据,而不仅仅是切分数据集。此外,作为一个闭集模型,SegVol分割未见类别的能力有限。然而,我们仍然对这一限制持乐观态度。当前的框架允许以相同的格式直接添加新数据,即使新数据包含以前从未见过的类别。这意味着该模型可以继承所有以前的知识,并在新的领域继续学习。这种适应性和持续学习能力使SegVol成为医学图像分割领域的一个很有前途的工具。
我们主要将SegVol用于计算机断层扫描**(CT)数据**,因为它具有快速图像采集,清晰细节和高对比度分辨率等优点。CT也是评估实体瘤的首选方法。然而,SegVol的灵活架构允许它与各种类型的体积医学图像兼容,比如MRI。我们相信这种多功能性可以使SegVol在体积医学图像的表示和分析中得到广泛应用。
有任何问题欢迎联系:438285719@qq.com 共同学习交流
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)